Aviäres expressionssystem für fremdproteine
Die
Erfindung bezieht sich auf die Verwendung einer Vogel-Zelle für die Herstellung
eines exogenen Proteins von Interesse in einem Tier, welches zu
der Spezies der Vögel
gehört,
wobei die Zelle durch einen Expressionsvektor transformiert ist,
welcher das das Protein kodierende Gen umfasst, wobei die Zelle
in die Keimhöhle
eines Embryos oder in den Blutkreislauf des Embryos eingeführt wird. Traditionell
sind die ersten pharmazeutischen Wirkstoffe durch Extraktion aus
Geweben unterschiedlicher Herkunft, wie Pflanzen, Tiere, wie sogar
Menschen, erhalten worden. Drei Hauptfaktoren haben zu der Entwicklung
von Ersatzmöglichkeiten
zu dieser Extraktionstechnik beigetragen. Die Verknappung der Gewebe aufgrund
der Zunahme der Nachfrage, die den tierischen und menschlichen Produkten
innewohnenden Risiken haben mit dem Auftreten oder der Identifizierung
von neuen Pathologien, insbesondere viralen und nichtkonventionellen,
zugenommen. Schließlich
und vor allen Dingen haben die Forschung und die Ausnutzung der Entdeckungen
der verschiedenen Zweige der Biologie (Biochemie, Molekularbiologie...)
es erlaubt, neue Moleküle
zu entdecken und folglich den ständig
wachsenden Bedarf an neuen Molekülen
zu befriedigen. Um
der Nachfrage an therapeutischen Produkten zu entsprechen, sind
die Expressionssysteme von Molekülen,
wie Proteinen mit therapeutischer Verwendung, mit einer aktiven
biologischen Komponente (Bakterium, Hefe, höhere eukaryotische Zelle...)
und einem genetischen Element, das die für die Produktion erforderliche
Unterstützung
beiträgt,
kombiniert worden. Die verschiedenen eingesetzten genetischen Träger sind zumeist
entweder einfache Expressionsvektoren, die für die Expression optimiert
worden sind, deren Integration aber weiterhin zufallsgesteuert bleibt,
oder virale oder retrovirale Systeme. Diese Letzteren erlauben nicht mehr,
weder den Ort noch das Niveau der Expression zu steuern. Diese
Herstellungsverfahren erleben gegenwärtig eine neue sowohl numerische
als auch qualitative Revolution. So hat im Zeitraum von einigen
Jahren dank der konstanten, schnellen Fortschritte der Genomik und
der Molekularbiologie die Anzahl von Molekülen, die ein oder mehrere therapeutische
und medizinische Potentiale aufweisen, signifikant zugenommen. Beispielsweise
repräsentieren
die Antikörper
mit therapeutischer Zielrichtung bereits etwa 50% von diesen Molekülen. Außerdem hat
sich die Komplexität
dieser rekombinanten Moleküle
erhöht.
Von einfachen Peptiden oder kleinen Zytokinen, die in Bakterien
leicht hergestellt werden, gelangt man fortschreitend zu komplexen
Molekülen
mit einer biologischen Aktivität,
die strikt von deren Sekundärstruktur,
von deren Faltung und von komplexen posttranslationalen Modifizierungen
abhängig
ist. Folglich reicht das einfache Mittel einer Herstellung in Bakterien
nicht mehr aus. Es
sind dann anspruchsvollere Techniken aufgetaucht, unter denen man
die in vitro-Kultur
von eukaryotischen Zellen, Hefen und Insektenzellen aufführen kann.
Vor noch kürzerer
Zeit sind die Fortschritte, die im Bereich der Herstellung von transgenen
Pflanzen und Tieren erzielt worden sind, mit Erfolg eingesetzt worden,
um diese komplexen Moleküle
entweder in Blätter
oder Samenkörner
von Pflanzenspezies (Tabak, Mais...) oder in den physiologischen
Flüssigkeiten
von Tieren, wie der Milch von Tieren (Ziege, Kuh, Sau, Kaninchen...),
herzustellen. Zudem
ist die Korrelation zwischen der Wirksamkeit des zielgerichteten
Ansteuerns („Targeting”) eines gegebenen
Genorts und dessen Transkriptionsniveau, insbesondere auf der Ebene
der durch den Vektor zielgerichtet angesteuerten Zellen, darunter
Stammzellen, nicht absolut. So weisen diese Methoden noch stets Probleme,
wie die Herstellungskosten, die Menge der erhaltenen Produkte und
technische Schwierigkeiten, wenn es sich darum handelt, komplexe
Moleküle
herzustellen, auf. Um
diese Probleme zu lösen,
wird im Rahmen der Erfindung vorgeschlagen, therapeutische Moleküle herzustellen,
indem die Expression zielgerichtet in speziellen Geweben, insbesondere
im Ei und im Speziellen im Eiweiß, erfolgt mit Hilfe von Tieren,
die modifiziert sind mit Stammzellen, die ihrerseits durch Rekombinationsvektoren
modifiziert sind. Die
verschiedenen Schritte, die für
das Ausführen
der Erfindung erforderlich sind, implizieren zuallererst die Konstruktion
von Vektoren, die das zielgerichtete Ansteuern („Targeting”) des gewählten Genorts in einer Vogel-Zelle
und insbesondere einer Stammzelle erlaubt, die Gewinnung von rekombinanten
Zellen, die diesen Vektor in dem gewählten Genort integriert aufweisen,
und die Erzeugung von mit diesen Zellen modifizierten Tieren. Einer
der Vorteile der Gesamtheit dieses Systems ist der gesteuerte Charakter
der Expression des Proteins von Interesse. Tatsächlich erlaubt die Wirkung
der substitutiven Ersetzung eines endogenen Vogel-Genorts durch
ein exogenes Gen oder Genfragment von Interesse, eine Produktion
des eingeführten
exogenen Proteins an Ort und Stelle des endogenen Genorts zu erhalten.
Indem beispielsweise in den Genort des Ovalbumins das zu produzierende
Gen durch die Gesamtheit des beschriebenen Mechanismus eingeführt wird,
wird das Produkt von diesem Gen gemäß dem Expressionsprofil des
Ovalbumins, das perfekt bekannt ist, produziert werden. So liefert
die Erfindung eine Lösung
für die
Probleme, die durch die zufällige
Integration einer heterologen Sequenz, deren Expression von ihrer
Chromatin-Umgebung abhängen
könnte,
aufgeworfen werden. Außerdem und
im Gegensatz zu den oben erwähnten
Methoden erlaubt das Vogel-Produktionssystem der
Erfindung, höhere
Mengen von aktiven Substanzen bei geringeren Kosten zu erhalten
angesichts dessen, dass sie leicht isoliert und gereinigt werden
können. Ein
Rekombinationsverfahren in einer Linie von lymphoiden Zellen (der
Linie DT40) ist dank eines Rekombinationsverfahrens aufgrund einer
besonderen Mutation (die Mutation wirkt sich auf das Expressionsniveau
des Proteins Rad54 aus) ermöglicht
worden (Kim et al., 1990; Baba et al., 1988; Buerstedde et al.,
1990; Buerstedde und Takeda, 1991; Bezzubova et al., 1993; Bezzubova
et al., 1997). In dieser Veröffentlichung
geben die Autoren zu, dass sie nicht in der Lage gewesen waren,
eine homologe Rekombination mit ES-Zellen auszuführen. Das
Dokument Das
Dokument von Prelle, K., et al., (1999) in „Cells, Tissues and Organs” (ISSN:
1422–6405),
Band 165, Seiten 220–236,
beschreibt bezüglich
embryonaler Vogel-Stammzellen,
dass diese die einzigen embryonalen Stammzellen neben den embryonalen
Stammzellen von der Maus sind, bei denen die Transmission in der
Keimbahn gezeigt worden ist. Im
Gegensatz zu dieser Lehre hat der Anmelder gezeigt, dass ein Verfahren
zur zielgerichteten homologen Rekombination eine Expression eines
exogenen Moleküls
von Interesse in Vogel-Systemen erlaubt und dass genau dieses Verfahren
auf der Versuchsebene in verschiedenen Arten von Vogel-Zellen, darunter
Linien, primäre
Zellen und Stammzellen und embryonale Stammzellen, bewältigbar
ist. Der
Gegenstand der Erfindung ist folglich: Die
Gesamtheit dieser Erfindung erlaubt folglich, ein neues System zur
Herstellung von Molekülen
von Interesse gemäß einer
perfekt vorhersagbaren, denn sowohl auf räumlicher wie zeitlicher Ebene
gesteuerten Weise vorzuschlagen, wodurch die Antworten auf die im
Stand der Technik existierenden Probleme gegeben werden. So
bezieht sich unter einem ersten Aspekt die Erfindung auf die Verwendung
einer Vogel-Zelle für
die Herstellung eines exogenen Proteins von Interesse in einem Tier,
das zu der Spezies der Vögel
gehört,
dadurch gekennzeichnet, dass die Zelle durch einen Expressionsvektor,
welcher das das Protein kodierende Gen umfasst, transformiert ist,
wobei die Zelle entweder in die Keimhöhle eines Embryos oder in den
Blutkreislauf des Embryos eingeführt
wird. Unter
Vogel-Zelle versteht man eine embryonale Stammzelle (ES), einen
zellulären
Klon der vorerwähnten
Zellen, eine ausgehend von den vorerwähnten Zellen etablierte Linie
oder einen „embryoid
body”,
der ausgehend von den vorerwähnten
Zellen erhalten worden ist. Die
verschiedenen Zellen, die zuvor ins Auge gefasst worden sind, werden
nachfolgend eingehender definiert: Der
für diesen
Zweck eingesetzte Vektor, ein Rekombinationsvektor, erlaubt eine
gewebespezifische Expression insbesondere im Eileiter (Legedarm),
der Leber, dem Blut, dem Mark und den lymphatischen Organen. Der
homologe Rekombinationsvektor (eine homologe Rekombination ermöglichende
Vektor) erlaubt, einen endogenen Genort zielgerichtet anzusteuern,
um ein Gen von Interesse an Ort und Stelle dieses endogenen Genorts
zu exprimieren. Auf der Ebene des Chromatins ist die homologe Rekombination
ein Verfahren, das darin besteht, partiell oder vollständig einen
Teil eines Gens durch ein Konstrukt zu ersetzen, das teilweise oder
in der Gesamtheit bestimmte Sequenzen, die mit eben diesem Gen identisch
sind, in sich aufnimmt. Die identischen Abschnitte erlauben, den
Austausch der DNA, die sich in dem endogenen Genort befindet, zu
realisieren und die modifizierten Teile in die betreffende und folglich
bewahrte Umgebung des zielgerichtet angesteuerten Genorts einzuführen. Daraus
resultiert eine Insertion von Modifizierungen, die sich in bestimmten Abschnitten
des Konstrukts befinden, in den zielgerichtet angesteuerten Genort.
Die verschiedenen Abschnitte des Konstrukts erlauben, die Expression
der eingeführten
Kassetten zu steuern. Diese Kassetten sind entweder abhängig von
ihrem eigenen Promotor oder abhängig
von der transkriptionellen Aktivität des zielgerichtet angesteuerten
Genorts, wenn die Kassette unter die Abhängigkeit von dem internen Promotor
platziert ist. Die Rekombinationsreaktion kann in drei große Schritte
unterteilt werden: Ein
jegliches Element, das in dem System vorhanden ist oder auf stabile
oder vorübergehende
Weise hinzugefügt
worden ist, das diese verschiedenen Mechanismen steuert, modifiziert
oder beeinflusst, wird einen essentiellen Beitrag in dem Rekombinationsprozess
und folglich hinsichtlich der Integrationseffizienz des Vektors
leisten. Im
Rahmen der Erfindung ist der Rekombinationsvektor, der erlaubt,
die Moleküle
in dem Vogel-System, insbesondere im Huhn, herzustellen, nicht ein
einfacher Zerstörungsvektor,
sondern ein substitutiver Ersetzungsvektor. Diese komplexen Vektoren
sind das Ergebnis der geordneten und aufeinanderfolgenden Kombination
von wenigstens einem der folgenden Elemente in einem klassischen
Klonierungsplasmid (pBSK, pUC, ...): Unter
cDNA versteht man die Gesamtheit oder einen Teil einer ein Protein
von Interesse kodierenden Nukleinsäuresequenz. Unter Protein von
Interesse versteht man ein Protein, ein Fragment eines Proteins
oder ein Peptid, das einen therapeutischen Nutzen für den Mensch
oder das Tier aufweist, aber auch eine jegliche Proteinform, die
dem Menschen oder dem Tier eine Wohltat spenden kann, was seinen
körperlichen
Status, seine Gesundheit, sein Verhalten oder seine Vitalität angeht,
was insbesondere ein endogenes Protein oder Peptid von Interesse
und ein exogenes Protein oder Peptid von Interesse, das nachfolgend
definiert wird, umfasst: Unter
exogenem Protein von Interesse versteht man ein jegliches Protein
von zu dem System exogener Herkunft, das in einem normalen physiologischen
Zustand in dem System, insbesondere dem Ei, nicht identifiziert
wird. Sein Vorhandensein ist das direkte oder indirekte Ergebnis
der genetischen und/oder biochemischen Modifizierung, die durch
den Vektor in dem System induziert wird. Die
hauptsächlichen
Merkmale eines Rekombinationsvektors, wie sie bis zum heutigen Tage
in Säugetier-Systemen
beschrieben worden sind, beruhen auf der Aufeinanderfolge der verschiedenen
Elemente innerhalb des Plasmids und auf der Positionierung der genomischen
Arme (Herkunft der DNA, Größe der homologen
genomischen Elemente, relative Dissymetrie...). In der Hauptzahl
der Fälle
ist die in vitro beobachtete Rekombinationsfrequenz abhängig von
dem zielgerichtet angesteuerten Genort und folglich der zielgerichtet angesteuerten
Chromatin-Umgebung (Ramirez-Solis et al., 1993; Hasty et al., 1995;
Hasty et al., 1991). Diese Umgebung ist eng verbunden mit der Physiologie
der Zelle. Es erweist sich, dass der auf die transfizierten Zellen
ausgeübte
Selektionsdruck eine Anzahl von hauptsächlich nicht-homologen Integrationen
bezogen auf die Anzahl von homologen Integrationen erlaubt. Das
Screeningverfahren, von dem Prinzip und Beispiele im Text erläutert werden,
erlaubt, die beiden Arten von Ereignissen zu unterscheiden. Das
Kennen, Evaluieren und Beherrschen des Anteils von homologer Integration
ist essentiell für
eine zufriedenstellende Expressionsstrategie. Die Zuverlässigkeit
der Rekombination zwischen den hinzugefügten Fragmenten und jenen des
Ursprungsmaterials muss gleichfalls die bestmögliche sein. Dies scheint zumindest
für die
ES-Zellen von der Maus und die humanen Fibroblasten der Fall zu
sein (Zheng et al., 1991; Lederman et al., 2000; Templeton, 2000). Außerdem kann
allein die Basenzusammensetzung eines Genorts die Rekombinationseffizienzen stark
beeinflussen, insbesondere durch das starke Vorhandensein von zahlreichen
CpG-Inseln (Yanez und Porter, 2000). Andere kryptische Elemente
(Poison-Sequenz)
können
gleichfalls die Effizienz des homologen Rekombinationsprozesses
modifizieren (Replikationsstartstellen, wiederholte Sequenzen...).
Außerdem scheint
die Frequenz der homologen Rekombination sehr stark von der Zielzelle
abhängig
zu sein mit besonderen Situationen, wie für die Linie von lymphoiden
Zellen DT40 vom Huhn, die beeindruckende Rekombinationsraten (> 10%) aufweist. Diese
Spezifität
ist mit einer Überexpression
des Moleküls
Rad54 verbunden, die zweifelsohne mit einer noch nicht identifizierten
Mutation verbunden ist, die in die Kontrolle des Regulationsniveaus
dieses Gens eingreift (Bezzubova et al., 1993; Bezzubova et al.,
1994; Bezzubova et al., 1997). Diese Beobachtung kann zu einer Erhöhung der
Rekombinationsfrequenz führen. Im
Rahmen der Erfindung ist die Effizienz der homologen Rekombination
in embryonalen Zellen von Vögeln
und bestimmten anderen primären
Vogel-Zellen, die in Form einer Linie etabliert worden sind, gezeigt worden. In
einer ergänzenden
Ausführungsweise
kann man Rad54 oder ein Protein der rad-Familie überexprimieren lassen mit dem
Ziel, die Effizienz des Rekombinationsprozesses zu verbessern. Einige
Untersuchungen, die in embryonalen Stammzellen von der Maus an verschiedenen
Genorten ausgeführt
worden sind, zeigen an, dass eine minimale Länge der 5' und 3' gelegenen Homologie-Sequenzabschnitte
erforderlich ist, um eine Rekombination an dem Genort mit einer
zufriedenstellenden Frequenz sicherzustellen (Hasty et al., 1991;
Thompson et al., 1989; Thomas und Capecchi, 1987). Wenn eine minimale Größe von 250–500 bp
ausreichend zu sein scheint, um ein Rekombinationsereignis von einer
der beiden Seiten sicherzustellen, bestünde der Schlüsselparameter
mehr in der gesamten Länge
der Homologie als in der jeweiligen Größe der Homologie-Sequenzabschnitte
(Elliott et al., 2001; Philips und Calos, 1999; Fujitani et al., 1995).
Diese Grenze wäre
minimal 5 bis 6 kb. Die Extrapolation der Ergebnisse ausgehend von
den sehr vollständigen
Daten, die bei den Bakterien erhalten worden sind, könnte darauf
hinweisen, dass der limitierende Faktor bei der Rekombinationseffizienz
nicht auf der Ebene der Untersuchung der Homologien zwischen den durch
den Vektor zugeführten
Donor-DNA-Fragmenten und den Empfänger-DNA-Abschnitten des zielgerichtet
anzusteuernden Genorts liegt (Yancey-Wrona und Caerini-Otero, 1995),
sondern vielmehr in dem Austauschsystem zwischen diesen Fragmenten,
das auf die Paarbildung folgt. Gleichwohl muss man große Vorsicht
walten lassen, was die Interpretation dieser Ergebnisse und deren
Extrapolation in das eukaryotische System angeht, die möglicherweise
empfindlich sind hinsichtlich des Vorhandenseins von Stellen, welche
die Rekombination an bestimmten Genorten vereinfachen („hot spot”). Die
Genorte von Immunglobulinen scheinen in dieser Hinsicht sehr besonders
zu sein. Es wurden Ereignisse von homologer Integration eines Vektors,
der eine Homologie lediglich in dem 3'-Arm aufweist, beobachtet (Berinstein
et al., 1992). Außerdem
scheinen die Rekombinationen in den Genorten von Immunglobulinen
besonders zu sein aufgrund der Aktivität der Stellen, die durch die
Rekombinasen, die diesen Genen eigen sind, erkannt werden. Es könnten möglicherweise
bestimmte Interferenzen mit der Ausrüstung von diesen Zellen und
dem Rekombinationsverfahren beobachtet werden. So
ist der Vektor ein homologer Rekombinationsvektor, welcher Homologie
zu den Sequenzen eines gegebenen Genorts aufweisende 5'- und 3'-Sequenzabschnitte
bzw. -Arme aufweist. Der zielgerichtet angesteuerte Genort wird
unter dem Genort des Ovalbumins und des Lysozyms ausgewählt. Dieses
Vorhandensein eines bedeutenden homologen Anteils ist umso notwendiger,
je bedeutender die heterologen Segmente sind (Kumar und Simons,
1993). So erfährt
der isogene Ansatz (Te Riele et al., 1992) eine Stärkung durch
die Beobachtung einer Verringerung der Frequenz der homologen Rekombination im
Falle einer Unterbrechung eines langen Homologien aufweisenden Fragments
durch variable „Stretchs”, die mit
dem Vorhandensein von Polymorphismus eines Genorts verbunden sind
(Lukacsovich und Waldman, 1999). Aber das symmetrische Vorhandensein
von heterologen Segmenten, die strangaufwärts von den eigenen homologen
Elementen gelegen sind, kann gleichfalls die Niveaus von homologer
Integration des Vektors verstärken.
Dieses Vorhandensein könnte
die Homologien vor den durch Exonukleasen induzierten Abbau-Vorgängen schützen. Eine
jegliche zielgerichtete Induktion (beispielsweise durch eine Meganuklease)
eines doppelsträngigen
Schnitts, eines in der DNA der Zielzelle spezifischen und seltenen
Schnitts, erhöht
die Rekombinationsfrequenzen. Es könnte scheinen, dass diese Induktion
das Reparatursystem auslöst
und den Einbau der exogenen DNA vereinfacht (Bibikova et al., 2001;
Sargent et al., 2000; Donoho et al., 1998; Sargent et al., 1997; Brenneman
et al., 1996; Hasty et al., 1992b). Dieser Ansatz wird insbesondere
eingesetzt, indem eine Stelle eingeführt wird, die spezifisch durch
eine Meganuklease, insbesondere vom Typ 1-Sce1 oder einen anderen, erkannt
wird, um die Rekombinationsereignisse zu begünstigen (Sang et al., 2000;
Cohen-Tanoudji et al., 1998; Robine et al., 1998; Jasin et al,.
1996). Es
wurden Vergleichstests zwischen linearen und zirkulären Vektoren
ausgeführt.
In dem Mäuse-System
scheint es, dass die Linearisierung eine vorab erfolgende Notwendigkeit
ist, um die Frequenzen zu erhöhen.
Andere Abgabewirkungen haben manchmal einen Einfluss (Yanez und
Porter, 1999). Die
verschiedenen gegenwärtig
in den Vektoren vorhandenen Elemente sind auf konstitutive Weise aktiv,
hat das homologe Rekombinationsereignis einmal stattgefunden. Indessen
kann die Expression eines exogenen Proteins im Ei eine etwaige Toxizität zugleich
auf der Ebene des Gewebes, das die Produktion sicherstellen wird,
und gegebenenfalls auf der Ebene des Eis, dem Ort der letztendlichen
Anhäufung,
zur Folge haben. Diese Anhäufung
muss folglich sowohl auf zeitlicher als auch räumlicher Ebene induziert und
beendet werden können. Einer
der möglichen
Ansätze
besteht in der Verwendung von induzierbaren und konditionellen Systemen.
Unter induzierbarem System versteht man beispielsweise das als Tet
off/tet an bezeichnete System. Dieses Letztere wird aus zwei Elementen
gebildet: dem Donor und dem Operator. Das Donor-System umfasst seinerseits
ein genetisches Konstrukt, das von einem Tetracyclinresistenz-System,
das in einem bakteriellen Tn10-Transposon identifiziert worden ist,
abgeleitet ist. In Abwesenheit von Tetracyclin blockiert das konstitutiv exprimierte
Repressorprotein TetR die Transkription des Gens, wodurch die Resistenz
gegen Tetracyclin sichergestellt wird. In Gegenwart von Tetracyclin
kann sich der Repressor nicht mehr an die Kontrollsequenzen binden.
Dieses System ist erfolgreich eingesetzt worden, indem diese tetR-Sequenz
mit der transaktivierenden Domäne
des viralen Proteins VP16 fusioniert wurde (Gossens und Bujard,
1992), Unter
den anderen induzierbaren Systemen kann man die Fusionen von diesen
verschiedenen Akteuren (VP16, Gal4...) mit Kontrollproteinsequenzen,
wie bestimmten Rezeptoren für
die nukleären
Hormone, die mehr oder weniger modifiziert sind, um auf spezifische
Weise auf das Vorhandensein von Analoga von Hormonen (mutierter
ER, welcher Tamoxifen bindet (Metzger et al., 1995; Indra et al.,
1999), der Ecdyson-Rezeptor, der PR, der RU486 bindet...) anzusprechen,
aufführen. Unter
konditionellem System versteht man beispielsweise die Systeme, die
bestimmte Enzyme, wie bestimmte Rekombinasen, einsetzen. Diese Enzyme
erkennen bestimmte und spezifische kleine Sequenzen (loxP-Sequenzen
für das
Enzym CRE, die Sequenz FLP für
die FRT-Rekombinase...), die dann in das zu steuernde Konstrukt
inseriert werden. Die allgemeine Wirkungsweise besteht darin, eine
Deletion der zwischen diesen Sequenzen gelegenen Elemente während der
Wirkung der Rekombinase vorzunehmen. Die Einführung der Rekombinase erfolgt
auf verschiedenen Wegen (vorübergehende
Transfektion, stabile integrierte Expression...) und ihre Expression
konditioniert die Exzisionsreaktion auf der Höhe der eingeführten Sequenzen. Indem
das Vorhandensein von diesen Rekombinasen mit den durch die Rezeptoren
der nuklearen Hormone induzierbaren Systemen kombiniert wird, macht
man die Gesamtheit des konditionellen Systems induzierbar. So kann
man beispielsweise die CRE-Rekombinase mit der mutierten Form des
ER, welche gegenüber
Tamoxifen empfindlich ist, fusionieren. Dieses System ist mit Erfolg
in dem Mäuse-System
eingesetzt worden (Metzger und Chambon, 2001; Vallier et al., 2001).
Für das
Mäuse-System
sind andere Systeme beschrieben worden, wie FLT-EcDR (Sawicki et
al., 1998). Die
Herkunft der Grundelemente der Vektoren ist variabel. Die Gerüste der
hauptsächlichen
Plasmide sind von kommerzieller Herkunft. Sie erlauben eine leichte
Vermehrung der Konstrukte, die diese tragen, in einer bakteriellen,
gut beherrschten Umgebung, jener von E. coli, sogar wenn verschiedene
Stämme
eingesetzt werden. So werden die Plasmide pBSK, pMCS5, pCl Néo eingesetzt,
um eine allgemeine Grundlage für viele
intermediäre
oder endgültige
Vektoren zu sein. Die
positiven Selektionskassetten (eine positive Selektion ermöglichenden
Kassetten) werden gebildet: Die
negativen Selektionskassetten (eine negative Selektion ermöglichenden
Kassetten) werden gebildet: So
umfasst der homologe Rekombinationsvektor gemäß der Erfindung in einer Plasmid-Grundstruktur eine
Aneinanderreihung von wenigstens einem Element, welches nacheinander
ausgewählt
wird aus: Dieser
Vektor kann gleichfalls d) die das exogene Protein kodierende Sequenz
fusioniert an ihrem 5'-Ende
mit c) einer kurzen Intronsequenz, welche insbesondere die Sequenz
SEQ ID NO: 1 umfasst, ihrerseits fusioniert mit b) einer Sekretionssignalpeptidsequenz,
insbesondere der das Signalpeptid von Lysozym kodierenden Sequenz,
umfassend die Sequenz SEQ ID NO: 2, umfassen. Der
erfindungsgemäße Vektor
kann gleichfalls d) die das exogene Protein kodierende Sequenz fusioniert
an ihrem 3'-Ende
mit einer polyA-Sequenz umfassen. Der Vektor kann gleichfalls mindestens
eine IRES-Sequenz fusioniert mit wenigstens zwei das exogene Protein
von Interesse kodierenden Sequenzen oder wenigstens eine IRES-Sequenz
fusioniert mit wenigstens zwei Sequenzen, welche unterschiedliche
Ketten, welche ein Protein von Interesse bilden, kodieren, insbesondere
die schweren und leichten Ketten eines Antikörpers von beliebiger Natur,
insbesondere eines monoklonalen Antikörpers, eines fab-Fragments,
umfassen. Unter
den IRES wählt
man die IRES der Gruppe I oder der Gruppe II, insbesondere die Sequenzen V130
( Diese
Vektoren sind gekennzeichnet durch die aufeinanderfolgende Kombination
eines Promotors, einer cDNA oder eines Abschnitts eines Gens, einer
polyA-Sequenz, welche das Anhalten der Transkription erlaubt. Nach
Transfektion von eukaryotischen Zellen mit diesen Vektoren weisen
diese Vektoren den Vorteil und den Nachteil auf, dass sie sich zufällig in
das Genom integrieren. Die Effizienz ist oftmals gut und es kann
eine große
Anzahl von Integrationsstellen erhalten werden. Indessen macht der
zufällige
Charakter dieser Insertion die Expression ausgehend von dem Promotor,
den der Vektor enthält,
abhängig
von der Umgebung der Insertionsstelle (Methylierung, Imprinting,
Enhancer, Silcencer...). So
kann man unter den einsetzbaren Promotoren, die diesen Schwierigkeiten
entsprechen und mit einem Ziel einer gewebespezifischen Expression
eingesetzt werden, auf nicht erschöpfende Weise den Promotor des
Gens von Lysozym (von 2500 bis 100 bp), aber auch den Promotor des
Gens des Ovalbumins in langen Formen (von 5 bis 1 kb), enthaltend
positive und negative Regulationsregionen, oder kurzen Formen (von
1000 bis 100 bp), wissend, dass ein minimaler Promotor von etwa
100 bp durch verschiedene Hormone spezifisch aktiviert werden kann
(Monroe und Sanders, 2000), erwähnen. Das
nachfolgende Beispiel 1 (Vergleichsbeispiel) veranschaulicht die
Möglichkeiten
zur Gewinnung von Stammzellklonen mit solchen einfachen Expressionsvektoren. Der
Vektor kann folglich ein Expressionsvektor sein, welcher die das
Protein von Interesse kodierende Sequenz fusioniert mit wenigstens
einem Element, ausgewählt
aus Dieser
Expressionsvektor und der Rekombinationsvektor können eine IRES-Sequenz umfassen,
die mit wenigstens zwei das gleiche Protein von Interesse kodierenden
Sequenzen oder unterschiedliche Sequenzen kodierenden Sequenzen
fusioniert ist. Die
vorerwähnten
Vektoren erlauben, eine Vogel-Zelle, wie zuvor definiert, zu transformieren. In
vorteilhafter Weise ist die Zelle eine embryonale primäre Vogel-Zelle,
eine embryonale Vogel-Stammzelle, insbesondere die embryonalen Stammzellen,
die aus der Kultivierung von Blastodermen stammen. Die
embryonale Vogel-Zelle der Erfindung weist einen Phänotyp einer
alkalische Phosphatase-positiven embryonalen Stammzelle auf. Die
embryonalen Vogel-Zellen, die embryonalen Stammzellen und die embryonalen
Keimzellen sind dadurch gekennzeichnet, dass sie spezifisch mit
mindestens einem Antikörper,
ausgewählt
aus ECMA-7, SSEA-1, SSEA-3, TEC-01, EMA-1 und EMA-6, reagieren. In
einer besonderen Ausführungsweise
ist die Zelle eine Vogel-Zelle, die abgeleitet ist von embryonalen
Stammzellen, die induziert worden sind, so dass sie sich unter der
Einwirkung von verschiedenen Induktoren, insbesondere Retinsäure, Dimethylsulfoxid,
TPA, oder speziellen Kulturbedingungen differenzieren, insbesondere
durch die Bildung von „embryoid
bodies” (siehe
obige Definition). Alternativ
ist in diesen Vergleichsbeispielen die Zelle eine in Form einer
Linie etablierte Vogel-Zelle, insbesondere die hepatischen Zellen
LMH, die Monozytenzellen HD11 und die Fibroblastenzellen QT6. Wie
zuvor erwähnt,
kann die Zelle mit einem Expressionsvektor, welcher ein Protein
der Familie Rad, insbesondere das Protein Rad54 exprimiert, transformiert
sein. So
bezieht sich unter einem zweiten Aspekt die Erfindung auf ein Verfahren
zur Gewinnung einer durch einen der vorstehend definierten Vektoren
modifizierten Vogel-Zelle. Dieses
Verfahren kann die folgenden Schritte umfassen Diese
Selektion erfolgt vorzugsweise während
wenigstens etwa 2 bis 10 Tagen, insbesondere 2, 3, 4, 5 oder 6 Tagen. Die
Erfindung bezieht sich in vorteilhafter Weise auf ein Verfahren
zur Gewinnung einer embryonalen Stammzelle von Vögeln, die durch einen der vorstehend
definierten Vektoren modifiziert ist, welches die folgenden Schritte
umfasst: Unter
einem bestimmten Aspekt steuert der Rekombinationsvektor zielgerichtet
den Genort des Lysozyms an. Die
Erfindung bezieht sich gleichfalls auf ein zuvor erwähntes Verfahren,
das dadurch gekennzeichnet ist, dass der Kulturüberstand der rekombinierten
Klone das exogene Protein von Interesse enthält, insbesondere nach Induktion
des Klons mit Hilfe von verschiedenen Induktoren, insbesondere Retinsäure, Dimethylsulfoxid,
TPA, oder speziellen Kulturbedingungen, insbesondere durch die Bildung
von „embryoid
bodies”. In
diesem Verfahren sind die beiden Allele des zielgerichtet angesteuerten
Genorts vorzugsweise modifiziert. Außerdem sind
die Zellen embryonale Stammzellen von Vögeln, insbesondere die embryonalen Stammzellen,
die aus der Kultivierung von Blastodermen stammen. Diese Zellen
weisen einen Phänotyp
einer alkalische Phosphatase-positiven embryonalen Stammzelle auf. Die
embryonalen Vogel-Zellen des Verfahrens sind dadurch gekennzeichnet,
dass sie spezifisch mit wenigstens einem aus ECMA-7, SSEA-1, SSEA-3,
TEC-01, EMA-1 und EMA-6 ausgewählten
Antikörper
reagieren. Die
Erfindung bezieht sich gleichfalls auf ein vorstehend definiertes
Verfahren, in welchem die Zellen embryonale Stammzellen sind, die
induziert worden sind, so dass sie sich unter der Einwirkung von
verschiedenen Induktoren, insbesondere Retinsäure, Dimethylsulfoxid, TPA,
oder speziellen Kulturbedingungen, differenzieren, insbesondere
durch die Bildung von „embryoid
bodies”. Die
vorstehend definierten Zellen können
außerdem
mit einem Expressionsvektor, welcher ein Protein aus der Familie
Rad, insbesondere das Protein Rad54 exprimiert, transformiert sein. Für dieses
Verfahren kann das eingesetzte Medium Anti-Retinsäure-Antikörper (ARMA)
und ein Zytokin, ausgewählt
in der aus LIF, IL-11, IL-6 und deren verschiedenen Mischungen bestehenden
Gruppe, umfassen. Das
eingesetzte Medium kann gleichfalls verschiedene Faktoren, insbesondere
SCF, IGF-1, bFGF, CNTF und Oncostatin, umfassen. Unter
einem dritten Aspekt bezieht sich die Erfindung auf ein Verfahren
zur Gewinnung eines Tiers, das zu der Spezies der Vögel gehört, welches
in der Lage ist, ein exogenes Protein von Interesse zu exprimieren,
das dadurch gekennzeichnet ist, dass es die folgenden Schritte umfasst: Der
in dem Schritt a) eingesetzte Vektor erlaubt vorzugsweise eine gewebespezifische
Expression, insbesondere im Eileiter (Legedarm), in der Leber, dem
Blut, dem Knochenmark und den lymphatischen Organen. Die
Erfindung bezieht sich gleichfalls auf das vorerwähnte Verfahren,
um ein Tier zu erhalten, das zu der Spezies der Vögel gehört, welches
eine gewebespezifische Expression eines exogenen Proteins von Interesse
aufweist, welches dadurch gekennzeichnet ist, dass der Vektor ein
homologer Rekombinationsvektor ist, welcher unter verschiedenen
konstitutiven Elementen, die für
sein Funktionieren erforderlich sind, 5'- und 3'-Sequenzabschnitte mit Homologie zu
den Sequenzen eines Genorts, welcher unter dem Genort des Ovalbumins
und des Lysozyms ausgewählt
ist, aufweist. Ein
solcher Vektor kann die das exogene Protein kodierende Sequenz fusioniert
mit wenigstens einem Element, ausgewählt unter einer Intronsequenz,
einer Sekretionssignalpeptidsequenz, insbesondere dem Signalpeptid
des Lysozyms, welches die Sequenz SEQ ID NO: 2 umfasst, einer polyA-Sequenz,
einem IRES und einem Promotor, insbesondere ausgewählt unter
den Promotoren der Gene des Ovalbumins, der Ovomucoide, von Conalbumin
und von Lysozym, umfassen. Der
Schritt b) des Verfahrens, um ein Tier, das zu der Spezies der Vögel gehört, welches
eine gewebespezifische Expression eines exogenen Proteins von Interesse
aufweist, zu erhalten, kann außerdem
die Transformation der Vogel-Zellen mit einem Vektor, welcher ein
Protein aus der Rad-Familie, insbesondere Rad54, exprimiert, umfassen. Unter
einem ergänzenden
Aspekt bezieht sich die Erfindung auf ein Verfahren zur Herstellung
eines Proteins von Interesse, welches die Extraktion des in den
Geweben eines ausgehend von dem zuvor erläuterten Verfahren erhaltenen
Tiers exprimierten exogenen Proteins umfasst. In diesem Verfahren
wird das Protein vorzugsweise aus dem Blut, Eigelb oder Eiweiß extrahiert. Alternativ
kann das Verfahren zur Herstellung eines Proteins von Interesse
in der Extraktion des im Überstand
der aus dem erfindungsgemäßen Verfahren
hervorgehenden Zellen exprimierten exogenen Proteins bestehen. Die
Erfindung bezieht sich gleichfalls auf ein Tier, das zu der Spezies
der Vögel
gehört,
welches ausgehend von dem oben beschriebenen Verfahren erhalten
werden kann, welches dadurch gekennzeichnet ist, dass es ein exogenes
Protein in einem speziellen Gewebe, beispielsweise in der Leber,
dem Blut, dem Knochenmark, den lymphatischen Organen oder dem Eileiter
(Legedarm), exprimiert. Unter
einem anderen Aspekt bezieht sich die Erfindung auf ein Ei, welches
ausgehend von einem oben beschriebenen Tier erhalten werden kann,
das dadurch gekennzeichnet ist, dass ein Teil von diesen Komponenten,
insbesondere das Ovalbumin und das Lysozym, teilweise oder vollständig durch
ein exogenes Protein von Interesse, welches insbesondere unter den
Peptiden von therapeutischem Interesse, den Interleukinen, den Zytokinen,
den Hormonen und den Antikörpern
ausgewählt
wird, ersetzt ist. Das
Ei der Erfindung kann einen Anteil an exogenem Protein zwischen
einigen mg (1 bis 10 mg) und 500 mg Trockensubstanz anstelle eines
Teils oder der Gesamtheit von wenigstens einem endogenen Protein, welches
insbesondere unter dem Ovalbumin und dem Lysozym ausgewählt wird,
umfassen. Nachfolgend
werden nicht-einschränkende
Beispiele von Ausführungsweisen
der Erfindung angegeben. Die
Expression eines Proteins von Interesse in einer physiologischen
Flüssigkeit
eines Tiers, beispielsweise des Huhns, insbesondere im Ei und insbesondere
im Eiweiß erweist
sich als mit der Hilfe von verschiedenen molekularen Hilfsmitteln
(Tools), darunter den Expressionsvektoren, realisierbar. Das allgemeine Prinzip
der Erfindung besteht darin, direkt im Ei ein exogenes Molekül von Interesse
anstelle eines endogenen Moleküls
oder eines Teils dieses endogenen Moleküls exprimieren zu lassen. Das
Ei ist ein für
die Expression von exogenen Molekülen besonders gut angepasstes
Medium. Das Eiweiß ist
ein komplexes Medium, dessen biochemische Zusammensetzung ziemlich
gut charakterisiert ist (siehe nachfolgende Tabelle I): Arm
an Lipiden (0,02%), an anorganischen Ionen und an Gluciden (0,5%,
darunter freie Glucose), besteht das Eiweiß hauptsächlich zu 88% aus Wasser und
aus in Lösung
befindlichen Proteinen (11,5%). Die Proteine des Eiweiß sind in
ihrer Gänze
gut charakterisiert, wenn auch deren genaue Anzahl noch Schwankungen
unterliegt (Stevens, 1991; Li-Chan
und Nakai, 1989; Sauveur, 1988). Man einigt sich im Allgemeinen darauf,
dass etwa 40 unterschiedliche Proteine identifiziert worden sind.
Unter jenen unterscheidet man fünf hauptsächliche
Proteine (Ovalbumin, Conalbumin, Ovomucoid, Ovomucin a und b und
Lysozym), die ihrerseits allein nahezu 83–84% der Proteine des Eiweiß repräsentieren.
Angesichts dieser Zusammensetzung und eines mittleren Gewichts an
Trockensubstanz von etwa 6–7
g repräsentieren
diese hauptsächlichen
Proteine folglich das Äquivalent
von etwa 4,8–5,6
g. Die in geringerem Ausmaß vorhandenen,
aber signifikant nachweisbaren Proteine machen 5 bis 6% der gesamten
Proteine aus. Unter diesen in geringerem Ausmaß vorhandenen Komponenten kann
man das Avidin aufführen,
das in der Welt der Diagnostik für
seine sehr hohe Affinität
und seine sehr hohe Spezifität
gegenüber
Biotin sehr bekannt ist. Die anderen Komponenten werden in geringen,
von einem Präparat
zum anderen schwankenden Anteilen identifiziert und sind oftmals
sowohl auf biochemischer wie molekularer Ebene schlecht charakterisiert.
Die Anzahl von Untersuchungen, die diese betreffen, ist außerdem in Übereinstimmung
mit ihrer geringen Repräsentanz
wenig bedeutend. Die physikalischchemischen Eigenschaften der hauptsächlichen
Proteine des Eiweiß werden
aufgeführt
(siehe nachfolgende Tabelle 2). Die
Proteine des Eiweiß werden
allesamt durch die Zellen des Eileiters (Legedarms) auf der Höhe des Magnums
hergestellt. Es werden verschiedene Zellarten gefunden und zwischen
den Zellen, die für
die Sekretion verantwortlich sind, wird eine bestimmte Spezialisierung
beobachtet. Die kalkbildenden (calciformen) Epithelzellen (welche
gleichfalls als muköse
Zellen bezeichnet werden) sind auf die Produktion von Avidin und Ovomucin
spezialisiert, wohingegen die Zellen der tubulären Drüsen präferentiell Lysozym und Ovalbumin
sekretieren. Die Verteilung dieser verschiedenen Zellarten ist im
sekretorischen Epithel des Magnums variabel, es wird aber keinerlei
Regionalisierung beobachtet (Sauveur, 1988). Die
Synthese der Proteine des Eiweiß erfolgt
auf der Ebene der Zellen kontinuierlich. Die glandulären und
kalkbildenden Epithelzellen „lagern” die Proteine,
die während
des Transits in das Magnum in einigen Stunden (etwa 3 h 30) um das
Eigelb herum abgegeben werden. Die mechanische Deformation induziert
diese sehr schnelle Sekretion und Ablagerung (Sauveur, 1988). So
erscheint es für
den guten Ablauf der Sekretion der exogenen Proteine in das Eiweiß essentiell, alle
diese Regulationssignale auf molekularer und physikalischer Ebene
aufrechtzuerhalten. Auf
der Ebene eines chimären
Tiers ist die Voraussetzung für
eine jegliche Produktion eines exogenen Moleküls von Interesse das Vorhandensein
der genetischen Modifizierung im Eileiter (Legedarm) und insbesondere
in den Zellen des Magnums. Der privilegierte Ansatz des Einsatzes
von embryonalen Stammzellen scheint diesen Mosaik-Charakter zu erlauben.
Auf der Ebene der Etablierung einer Linie von Tieren wird dieser Beitrag
durch die adäquaten
Kreuzungen automatisch auf den heterozygoten, dann homozygoten Zustand übertragen,
denn er ist auf der Ebene des Genoms integriert. Das
Eigelb besteht im Wesentlich aus einer Anhäufung von Lipiden (Tabelle
1 oben) in Form von Lipoproteinen. Es ist das Ergebnis einer engen
Assoziierung der beiden Hauptproteine, des Vitellins und des Vitellenins,
mit Phospholipiden und Triglyceriden. Die anderen Bestandteile (Cholesterol,
Vitamine und fettlösliche
Pigmente) sind in geringeren Mengen vorhanden. Im Gegensatz zu den
Proteinen des Eiweiß werden
alle Proteine und die Komponenten des Eigelbs durch die Leber synthetisiert
und über
den Blutkreislauf transportiert, um sich auf der Ebene des Eigelbs
während
der Entwicklung der Follikel anzuhäufen (Sauveur, 1988; Nau, 1987).
Man findet im Eigelb gleichfalls einen nicht vernachlässigbaren
Anteil von Immunglobulinen, die direkt im Verlauf der Herstellung
von diesem Vitellus angehäuft
werden. Das Vorhandensein von diesen Immunglobulinen mütterlicher
Herkunft stellt überdies
einen gewissen Immunschutz im Verlauf der ersten Tage, ja sogar
mehr, des Lebens des Kükens
sicher. Es wird gerade damit begonnen, den Transportmechanismus von
diesen Immunglobulinen und deren Anhäufung im Ei zu untersuchen.
Das Verfahren zur Reinigung dieser für den Vogel spezifischen IgY-Moleküle (Äquivalente
zu den IgG der Säugetiere)
wird gut beherrscht. Diese Eigenschaft kann mit Erfolg eingesetzt
werden, um Immunglobuline von Vögeln
in relativ bedeutender Menge (manchmal in der Größenordnung von 10 mg pro Ei)
zu erhalten, ja sogar die Anhäufung
davon durch eine zielgerichtete Immunisierung des Eier legenden
Tiers zu begünstigen.
Es ist außerdem
gezeigt worden, dass die humanen Immunglobuline sich gleichfalls
in dem Eigelb anhäufen
können
durch einen Mechanismus, der zu den endogenen Immunglobulinen ähnlich ist
(Mohammed et al., 1998). Unter dieser Perspektive, durch das Huhn
bestimmte Immunglobuline produzieren zu lassen, kann eine spezifische
Expressionsstrategie entwickelt werden. Der
Hauptteil der Gene, die die hauptsächlichen Proteine des Eiweiß spezifizieren,
sind auf molekularer Ebene untersucht worden. Die kodierenden Phasen
sind im Allgemeinen identifiziert und der Hauptteil der Sequenzen
der entsprechenden cDNAs findet sich in den Genbanken. Auf genomischer
Ebene sind die Fortschritte langsamer gewesen. Die Struktur der
Genorte des Hauptteils der hauptsächlichen Moleküle ist vollständig oder
partiell veröffentlicht
worden. Gleichwohl können
die veröffentlichten
Sequenzen sich als unvollständig
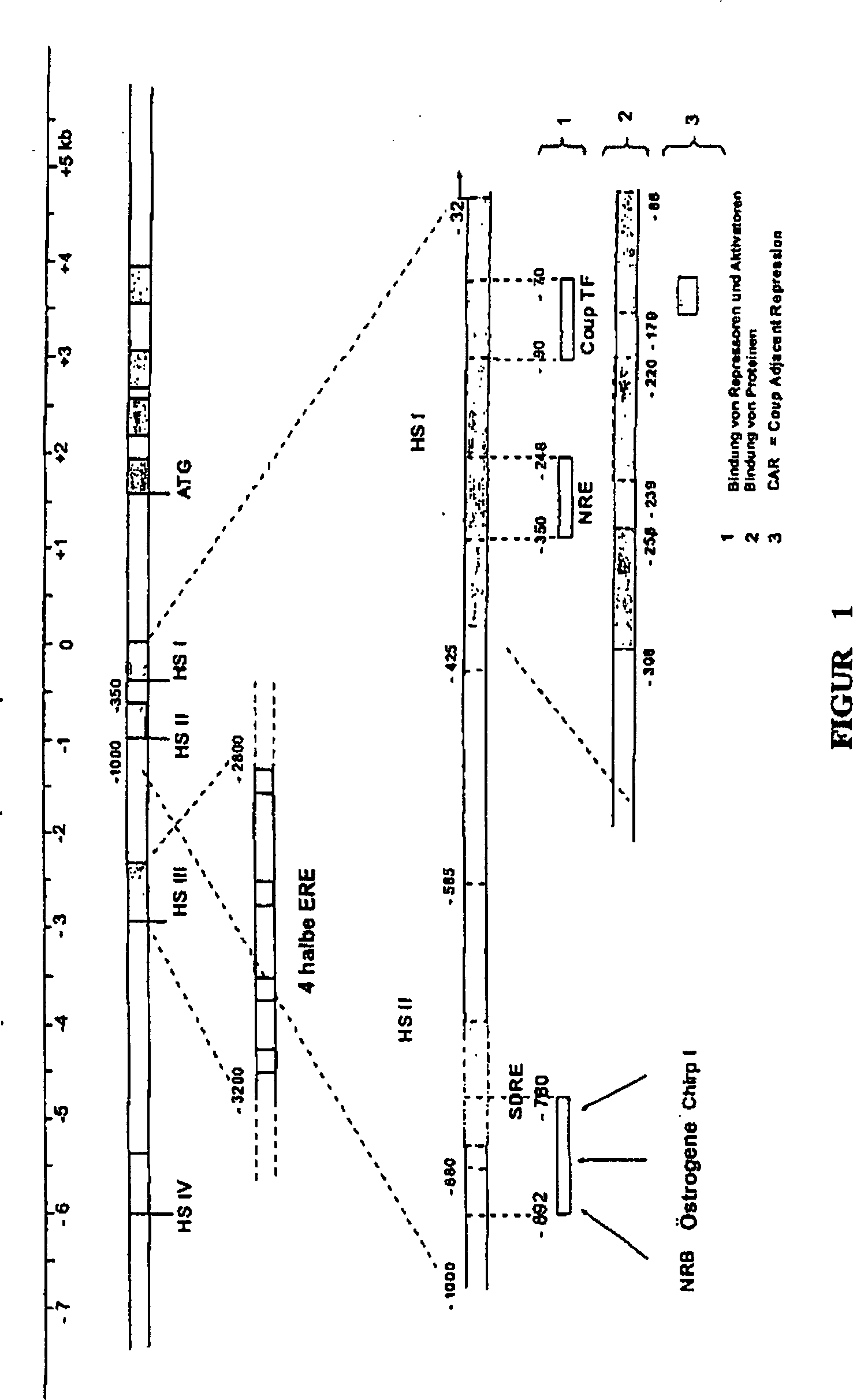
erweisen (siehe Tabelle 3 unten). Der
Genort des Ovalbumins mit einer Größe von 100 kb gehört zu der
Multigen-Familie
der Serpine und enthält
3 verwandte Gene, ova, ovaX und ovaY. Sie werden allesamt im Eileiter
(Legedarm) eines im Zustand des Eierlegens befindlichen Huhns exprimiert
(LeMeur et al., 1981; Baldacci et al., 1981). Durch elektronenmikroskopische
Untersuchungen strukturell seit 1979 bekannt (Gannon et al., 1979),
befindet sich der Genort des Ovalbumins auf dem Chromosom 2 (Dominguez-Steglich
et al., 1992). Die erste vollständige
Sequenz, die 1978 veröffentlicht
worden ist, ist jene des Ovalbumins. Seine prä-mRNA hat eine Größe von 7564 bp
und umfasst 7 Exons (McReynolds et al., 1978). Die gespleißte mRNA
enthält
1872 bp (Woo et al., 1981). Die
Transkription des Ovalbumin kodierenden Gens (wie überdies
jene des Conalbumins, siehe 2.3) wird durch die Steroidhormone gesteuert.
So stimuliert die Zugabe von Östrogenen
die Produktion von Ovalbumin um das 20-fache und jene des Conalbumins
um das 2,5-fache (N'guyen
et al., 1979). Vor einer hormonalen Stimulation sind die Transkripte
des Ovalbumins in den Zellen des Eileiters (Legedarms) nicht nachweisbar,
wohingegen sie proportional zu der Ostrogendosis mehr als 50% der
gesamten Transkripte einer Zelle nach maximaler Stimulation repräsentieren
(Dean et al., 1983). Während
das Transkriptionsniveau des Conalbumins zu der Menge der Ostrogenrezeptoren
im Kern direkt proportional ist, weist jenes des Ovalbumins darauf
hin, dass es mehrere Bindungsstellen der Östrogenrezeptoren auf der Ebene
des Promotors gibt (Palmiter et al., 1981). Die nukleosomale Struktur
der Gene des Ovalbumins und des Conalbumins modifiziert sich im
Verlauf der transkriptionellen Aktivierung. Die genomischen Fragmente
assoziieren sich mit der nuklearen Matrix und weisen vier Hauptregionen
von übermäßiger Empfindlichkeit
gegenüber
DNAse infolge einer Behandlung mit den Hormonen auf (Ciejek et al.,
1983). Diese Regionen befinden sich in der 5'-Region des Promotors und 3' von dem letzten
Exon auf der Höhe
der Polyadenylierungsstelle (Bellard et al., 1982; Bellard et al.,
1986) ( Die
Ergebnisse von zahlreichen Arbeiten, die mit dem Promotor des Ovalbumins
ausgeführt
worden sind, haben erlaubt, die verschiedenen Nukleotidregionen,
die das Ziel der Wirkung von diesen Hormonen und Faktoren sind,
aufzuklären
(Sensenbaugh und Sanders, 1999). Sukzessive Deletionsarbeiten auf
der Ebene des Promotors und Transfektionsexperimente von verschiedenen
Konstrukten zeigen, dass die Aktivierung des Promotors in den primären Zellen
des Eileiters (Legedarms) des Huhns in Gegenwart von Östrogenen oder
nicht optimal ist. Das Vorhandensein von gewebespezifischen Aktivatoren
und Repressoren ist folglich für
die Expression von diesem Gen essentiell (Dierich et al., 1987).
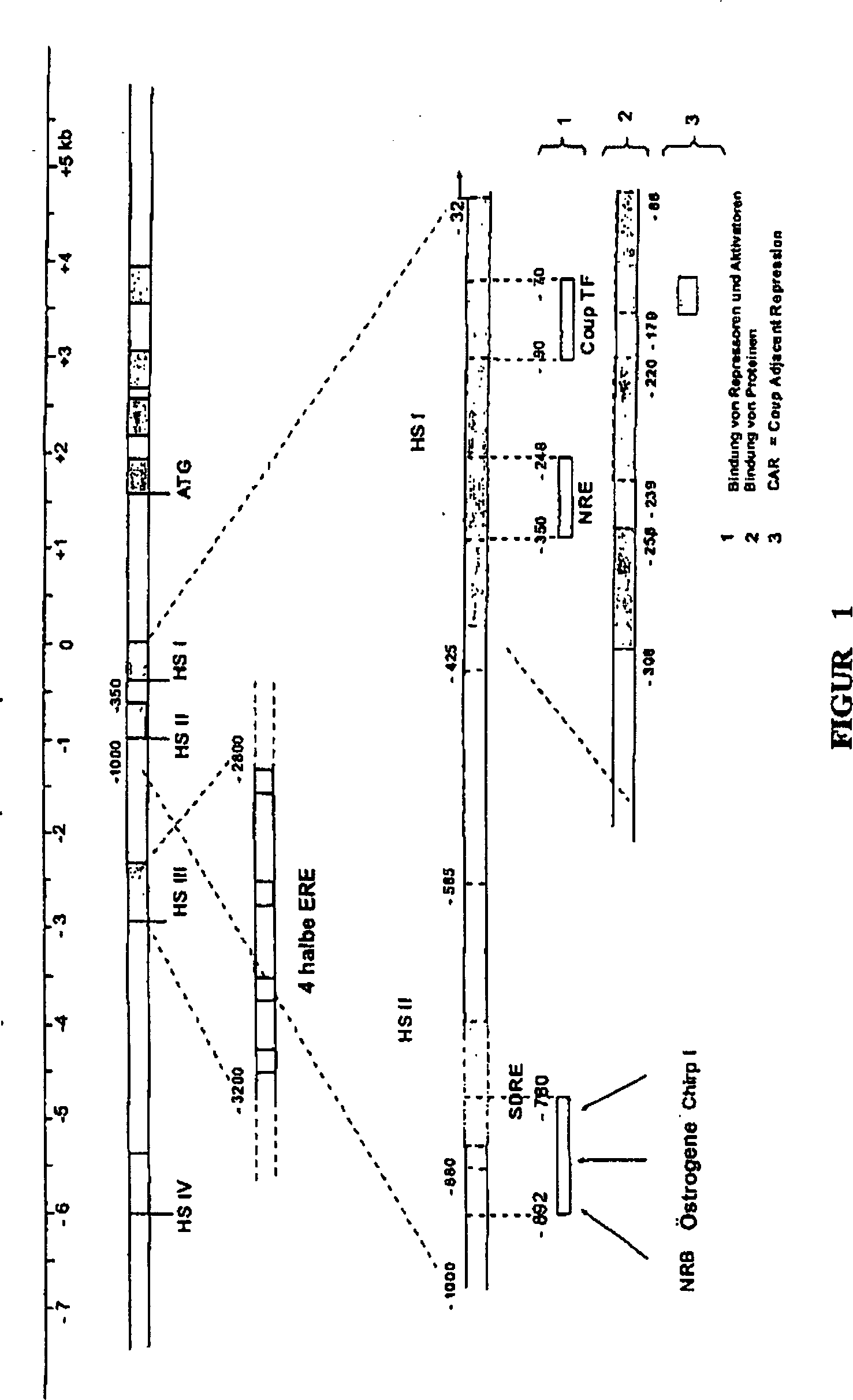
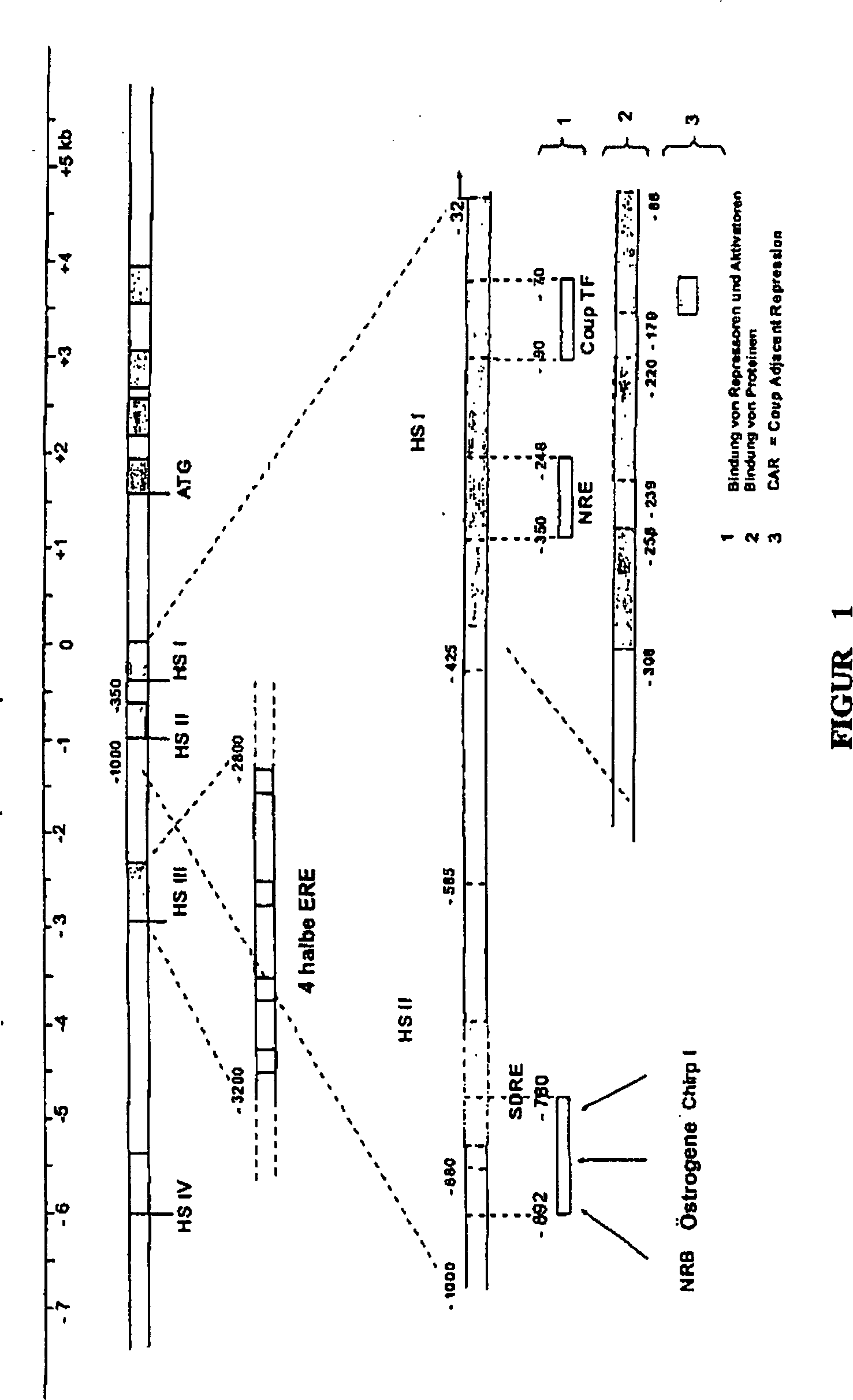
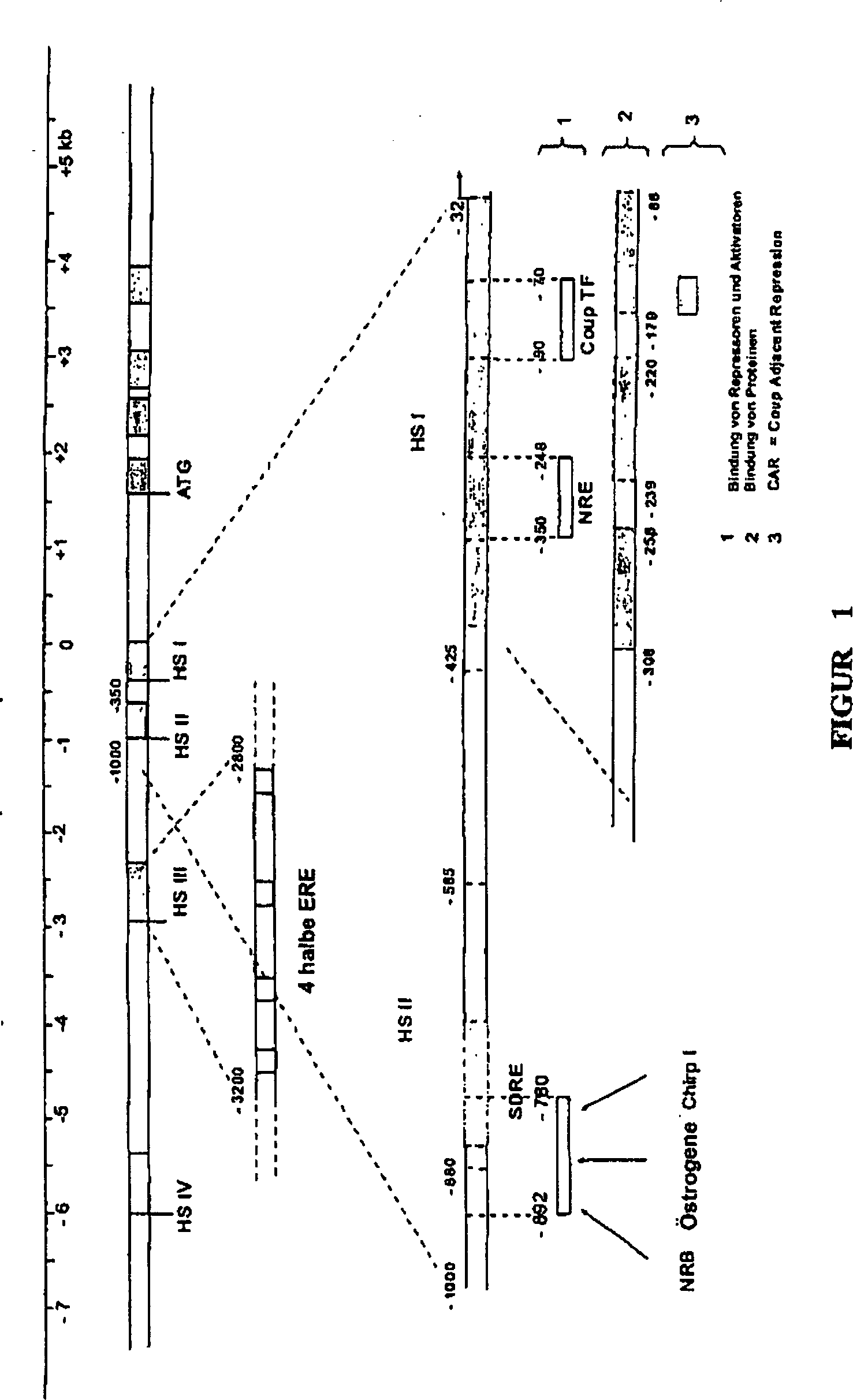
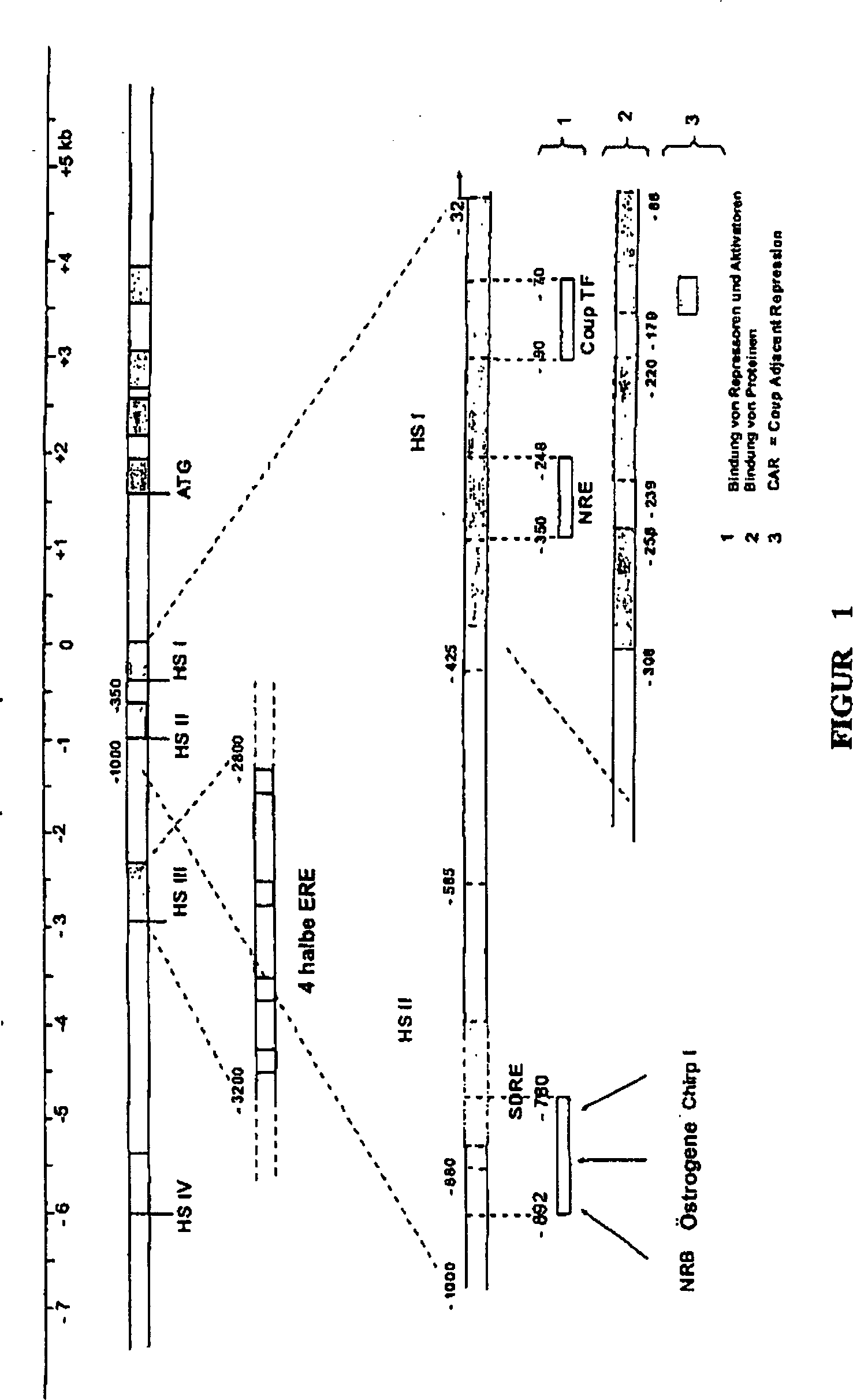
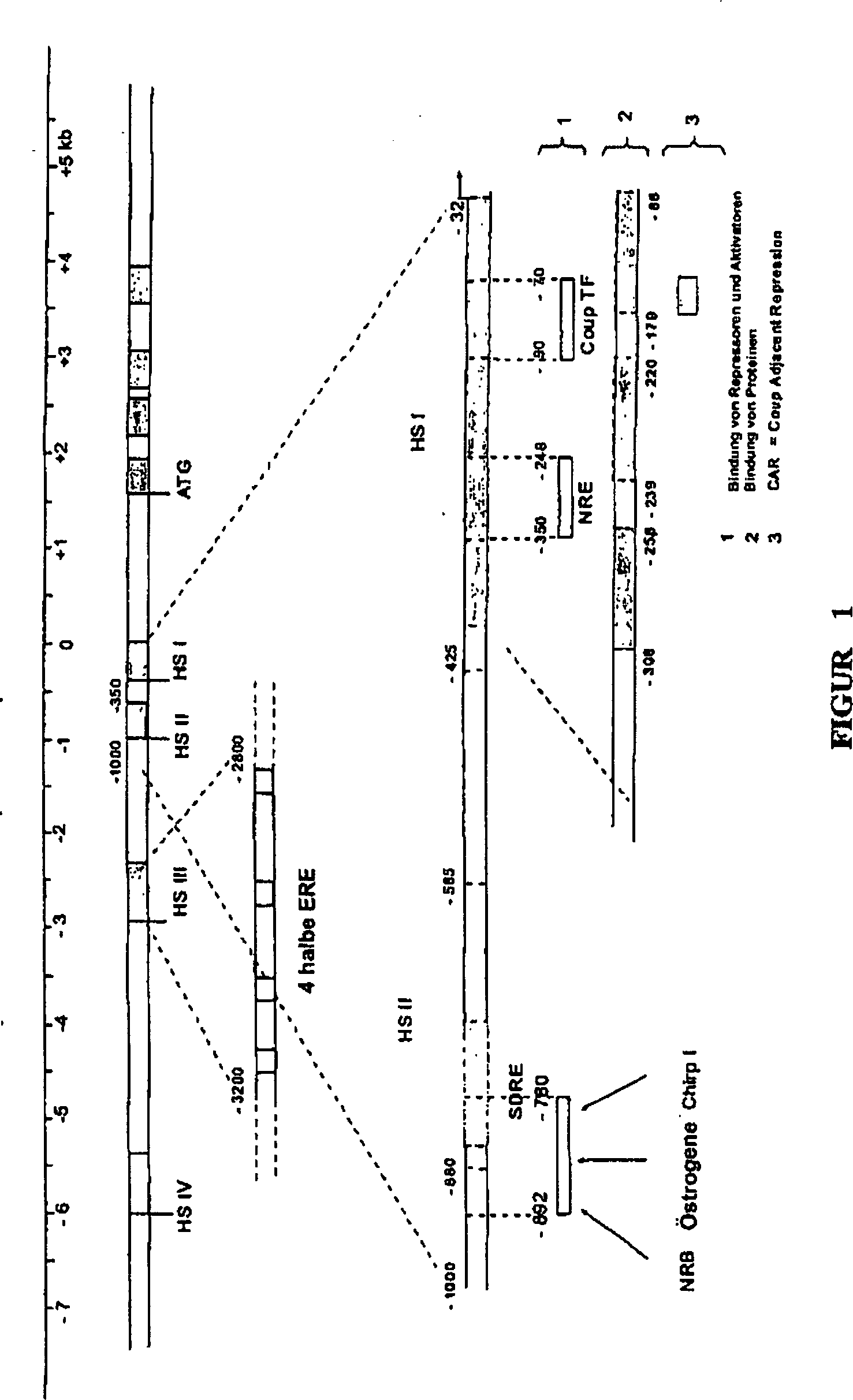
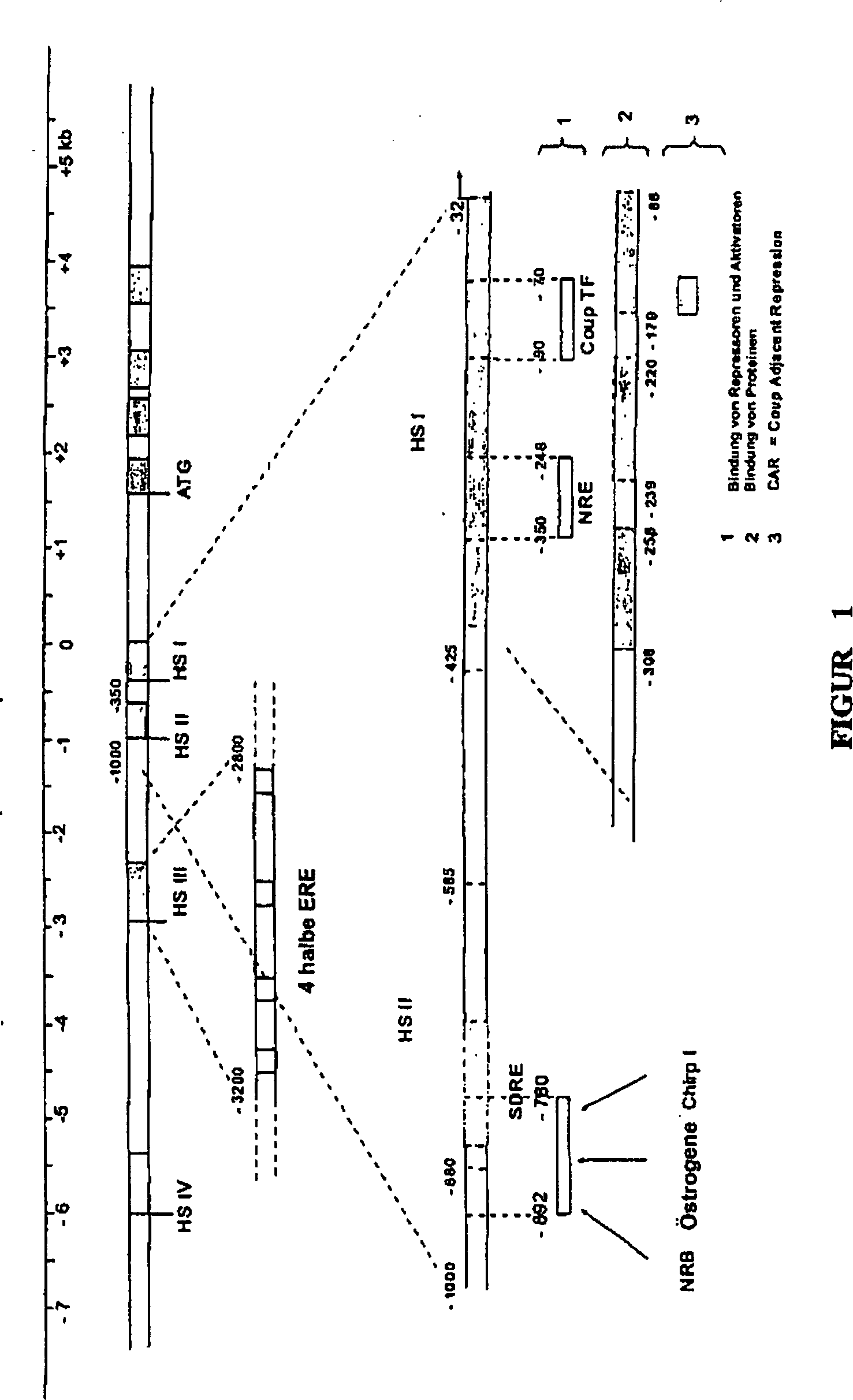
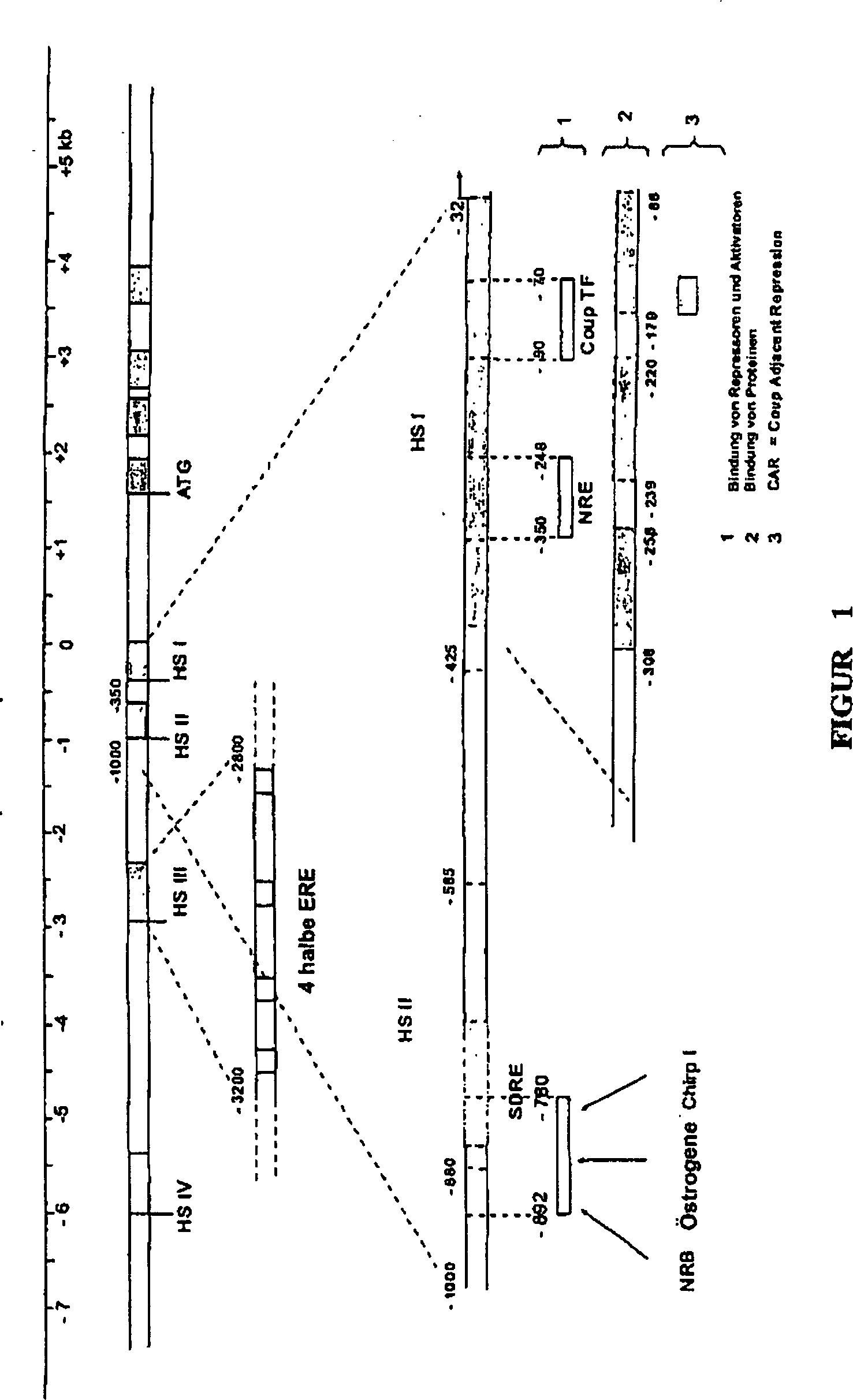
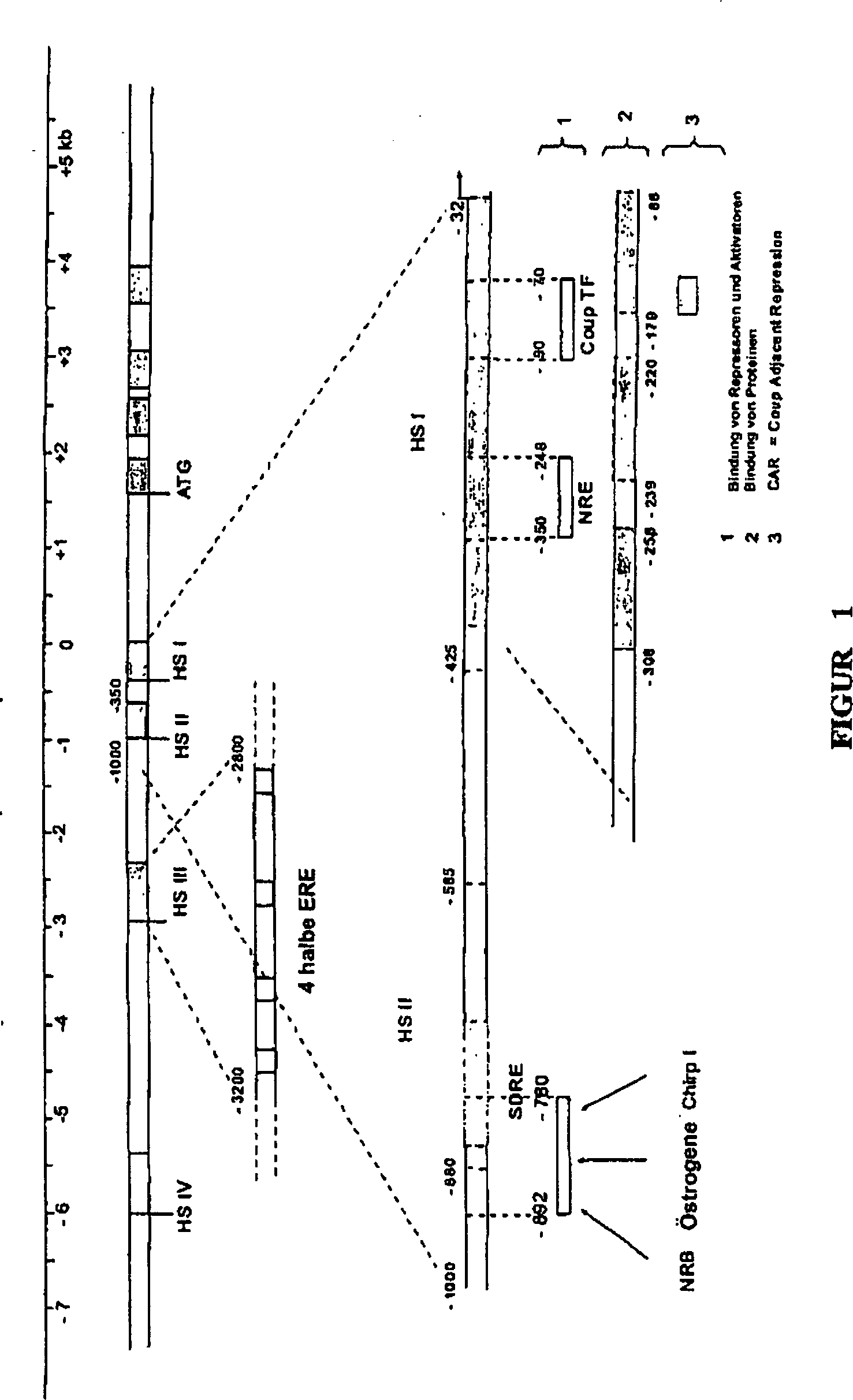
Die Man
findet so aufeinanderfolgend eine HSIV-Region im Bereich von –6000 bp
bezogen auf die Transkriptionsstartstelle, die keine auf Östrogene
ansprechenden Elemente aufweist. Eine Region HS III, die zwischen –3200/–2800 bp
bezogen auf die Transkriptionsstelle lokalisiert ist, umfasst die
hauptsächlichen
Regulationsstellen durch die Östrogene.
Die Deletion von dieser Region vernichtet den größten Teil der HSIII-Reaktion,
die durch vier wiederholte halbe ERE-Sequenzen, die voneinander
durch etwa 100 Nukleotide getrennt sind, vermittelt wird. Diese
Elemente kooperieren mit der Stelle der in der Nähe gelegenen TATA-Box. Jede Stelle
bindet einen Rezeptor schwach, aber die Kooperation der verschiedenen
Stellen stellt einen starken induzierbaren Charakter in einer Umgebung
eines Minimalpromotors oder heterologen Promotors sicher. Die gleiche
Region zeigt inhibitorische Elemente der Expression des Gens, Elemente,
die in den nicht-produktiven Zellen aktiv sind, aber in einer Situation
außerhalb
des Eileiters (Legedarms) (Kato et al., 1992; Muramatsu et al.,
1995). Die
HS II-Region umfasst das SDRE („Steroid Dependent Regulatory
Element”),
das zwischen –800 und –585 bp
bezogen auf die Transkriptionsstartstelle (+1) lokalisiert ist.
Diese Region weist Homologien zu dem Promotor des Gens NF-kappa
B auf, was Kontrollen durch gemeinsame Faktoren nahelegt (Schweers und
Sanders, 1991). Mehrere Faktoren aus der Familie der Proteine W-H,
die Proteine Chirp-I, Chirp II, Chirp III („Chicken ovalbumin Induced
Regulatory Protein I”)
binden sich gleichfalls an das SDRE einzig in Gegenwart von Östrogen
und von Glucocortidoiden (Dean et al., 1996; Dean et al., 1998;
Dean et al., 2001). Erneut führt,
wie um die Komplexität
der eingesetzten Regulationen zu zeigen, das alleinige Vorhandensein
von dieser SDRE-Region innerhalb eines heterologen Promotors nicht
zu einer von Steroidhormonen abhängigen
Reaktion (Response). Gleichwohl scheint diese Region von diesen
Hormonen abhängig
zu sein, wenn sie mit der HSI-Region assoziiert ist. Diese
Region HS I, die von –350
bis –32
lokalisiert ist, ist die NRE (Negative Response Element)-Region,
die die hauptsächliche
negative Regulation des Promotors kontrolliert. NRE reprimiert die
Expression des Gens in Abwesenheit von Steroidhormonen. Das Vorhandensein
allein von NRE oder allein von SDRE reicht nicht aus, um ein exogenes
Gen zu regulieren. Diese Sequenzen müssen kooperieren, um „die Derepression zu
induzieren” (Hasty
et al., 1994; Schweers et al., 1990; Sanders und McKnight, 1988).
Aber die Ansicht ist grob vereinfachend, denn diese HSI-Region wird
tatsächlich
aus einem veritablen Mosaik von Stellen, die eine positive und negative
Regulation der Transkription des Gens sicherstellen, gebildet. Es
sind drei Domänen
hinsichtlich des Einflusses von ihren Wirkungen unterscheidbar.
Domäne
I. Die Region –239
bis –220
ist eine Sequenz vom Silencer-Typ. Die drei Zonen weisen Affinität für spezifische
Proteinkomplexe der Zellen des Eileiters (Legedarms) auf, aber die
bedeutendsten sind die Regionen –280 bis –252 und –134 bis –88. Diese Region umfasst das
Motiv CAR („COUP
Adjacent Repression”).
Die als NRE bezeichnete Region ist tatsächlich komplexer als zu Anfang
beschrieben (Haecker et al., 1995). Es ist auch eine positive Regulationsstelle,
die den Faktor delta EF1 („Estrogen
responsive transcription factor”)
bindet, identifiziert worden. So überlappen sich die Wechselwirkungsstellen
(–197
bis –95)
des Progesterons und der Östrogene
partiell (Dean et al., 1983; Dean et al., 1984). Strangabwärts von
der Position von –95
bp haben ergänzende
Deletionen die beobachteten Konzentrationen der Transkripte dramatisch
abfallen lassen (Knoll et al., 1983). Außer den Östrogenrezeptoren ist ein Wichtiger
der Regulationsfaktoren der Orphelinrezeptor COUP TF („Chicken
Ovalbumin Upstream Promoter Transcription Factor”) (Sawaya et al., 1996; Robinson
et al., 1999). Dieser Faktor wird ubiquitär in allen Geweben exprimiert.
Durch gemeinsame Reinigung mit dem Östrogenrezeptor identifiziert,
weist er Affinität
zu einer zwischen –90
und –70
bp gelegenen Region auf (Wang et al., 1987; Hwung et al., 1988a, Hwung
et al. 1988b; Monroe und Sanders, 2000). Die Bildung von Rezeptor-Rezeptor-Komplexen
zwischen COUP-TF und den verschiedenen Rezeptoren für die nuklearen
Hormone (VDR, ER, TR, GR, PR, RXR, HNF-4...) auf der Ebene der verschiedenen
HRE, darunter ERE, begünstigt
die Bindungen an diese Regulationselemente. COUP-TF reguliert die
Wirkung von ER durch direkte Kontakte mit der DNA, aber auch über direkte
Protein-Protein-Wechselwirkungen
zwischen Rezeptoren (Klinge et al., 1997). Die Wirkungsweisen von COUP-TF
und verschiedenen verwandten Rezeptoren (COUP-TFII oder APRI) sind
komplex (Kimura et al., 1993) in einem positiven oder negativen
Sinne für
die Regulation der Transkription gemäß den im Spiel befindlichen
Proteinpartnern. Unter den beteiligten Proteinen kann man nicht
erschöpfend
ear 2 (Pereira et al., 1995); N-CoR („Nuclear Repressor Coreceptor”) und SMRT
(„Silencing
Mediator for retinoic acid and Thyroid hormone Receptor”) (Shibata
et al., 1997) aufführen. Der
Genort des Lysozyms ist ein Genort, der hinsichtlich der Größe stärker beschränkt ist
als jener des Ovalbumins. Er wird auf etwa 40 bp geschätzt (Sippel
et al., 1978; Short et al., 1996). Zwei MAR („Matrix Attachment Region”)-Einheiten,
die bei –11,1
kb/–8,85
kb und +1,3 kb bis +5,0 kb bezogen auf die Transkriptionsstartstelle
(+1) gelegen sind, begrenzen diesen Genort genauer auf 22 kb. Diese
MAR-Sequenzen bestehen aus mehreren Verankerungsstellen an die Proteinmatrix
der nuklearen Membran (Loc und Strätling, 1988; Phi-van und Strätling, 1996;
Phi-van, 1996). Das Hinzufügen
von MAR-Sequenzen zu heterologen Genen erhöht deren Transkription (Stief
et al., 1989; Phi-Van et al., 1990). Noch zusätzlich zu dem Vorhandensein
der MAR gibt es wenigstens eine Replikationsstartstelle der DNA
in dem Genort des Lysozyms, eine Startstelle, die mehrere Initiationsgabeln
der Replikation enthält
(Bonifer et al., 1997; Phi-van et al., 1998; Phi-van und Strätling, 1999).
Diese Stelle scheint überdies
den Genort des Lysozyms ganz und gar besonders zu machen hinsichtlich
der Klonierung, da bestimmte Abschnitte ziemlich schwierig in Klonierungsplasmide
subzuklonieren sind. Das Gen des Lysozyms beim Huhn umfasst 4 Exons
und weist eine bedeutende Homologie zu den Genen des humanen Lysozyms
und des Lysozyms von der Maus auf. Es wurden mehrere sekundäre Transkriptionsstartstellen
nachgewiesen, die im Allgemeinen in über die Gesamtheit des Genorts
verteilten wiederholten Sequenzen gelegen sind (Von Kries und Strätling, 1988).
Von –14
kb bis +6 kb, d. h. über
die vollständige
Gesamtheit des Genorts, wurden die Expression regulierende Sequenzen
identifiziert. Das Gen des Lysozyms wird in den reifen Makrophagen,
Zellen, in denen das Lysozym als Differenzierungsmarker eingesetzt wird,
konstitutiv exprimiert, wohingegen seine Expression in den Zellen
des Eileiters (Legedarms) unter die Kontrolle der Steroidhormone
gestellt ist (Short et al., 1996; Fritton et al., 1987; Jantzen
et al., 1986; Fritton et al., 1983). Diese Spezifitäten der
Expression sind von verschiedenen Sequenzen, von denen die hauptsächlichen
Stellen in der Man
unterscheidet zwei Kategorien von regulatorischen Sequenzen: die
sogenannten positiven Regulationssequenzen oder „Enhancer” und die sogenannten negativen
Regulationssequenzen oder „Silencer”. Diese
Sequenzen wurden nach der Analyse der elektrophoretischen Profile
der DNA, von Profilen, die nach Behandlung mit DNAse I erhalten
worden sind, als „hypersensible” Regionen
identifiziert. Die
hauptsächlichen
Enhancer- und Silencer-Stellen sind jeweils die Regionen E-6,1 kb, E-2,7 kb, E-0,2
kb bzw. N-2,4 kb, N-1,0 kb und N-0,25 kb. Bestimmte sind für myeloische
Zellen spezifisch (E-6,1 kb, E-2,7 kb, E-0,2 kb für die reifen
Makrophagen und N-2,4 kb in den Fibroblasten und unreifen Makrophagen) (Steiner
et al., 1987; Huber et al., 1995). In den Zellen des Eileiters (Legedarm)
stellt das Element E-6,1 kb den Hauptanteil der Spezifität sicher.
Die sequentielle Aktivierung der Gesamtheit von diesen unterschiedlichen
Stellen ist gleichfalls ein zell- und gewebespezifisches Regulationselement
(Regenhard et-al., 2001; Kontaraki et al., 2000). Detaillierter
besteht der Enhancer E-2,7 kb aus 4 Regionen (I bis IV). Die Region
I bindet den Faktor FEF („c-fes
Expression Factor”),
die Region II enthält
eine Erkennungsstelle für
den Faktor PU.1 aus der EST-Familie und ist spezifisch für Makrophagen
(Ahne und Strätling,
1994). Alle diese unterschiedlichen Regulationselemente überlappen
sich teilweise, zumindest in dem Gen vom Huhn, aber nicht auf der Ebene
des Gens von der Maus, einer Spezies, in welcher eine anzestrale
Verdoppelung das Gen und seine Regulationselemente voneinander getrennt
hat. Es wurden andere Elemente lokalisiert, insbesondere die Bindungsstellen
von ubiquitären
Transkriptionsfaktoren, wie NF-1, aber auch AP1, gelegen bei –0,2 kb
und –6,1 kb,
eine Stelle, für
welche eine Region von 157 bp die Spezifität der Expression in den Makrophagen
sicherstellt (Goethe et al., 1994; Theisen et al., 1986; Grewal
et al., 1992; Nowock und Sippel, 1982). Interessanterweise blockiert
das Onkogen vmyc (das beispielsweise in den HD11-Zellen vorhanden
ist), welches die Differenzierung der Monozyten zu Makrophagen inhibiert,
die Transkription, indem es die Bindung des Faktors C/EBP inhibiert
(hink et al., 1996). Die
Elemente (HRE), welche die Empfindlichkeit der Transkription gegenüber den
Steroidhormonen (PRE für
Progesteron und GRE für
Glucocorticoide) sicherstellen, sind in der bei -0,2 kb von der
Transkriptionsstartstelle (+1) des Promotors gelegenen Region vorhanden
(Renkawitz et al., 1982; Renkawitz et al., 1984a; Renkawitz et al.,
1984b; Hecht et al., 1988). Spezieller stellen zwei Stellen zwischen –220 und –140 bp und
zwischen –80
bp und –50
bp das Wesentliche der Regulation sicher (Altschmied et al., 1989;
Dolle und Strätling,
1990; Von der Ahe et al., 1986). Ein
bedeutender Teil der Regulation wird durch „Silencer” gesteuert. Zwei von jenen
(N-1,0 kb und N-0,25 kb) inhibieren die Transkription, sogar von
heterologen Promotoren (Baniahmad et al., 1987; Faust et al., 1999).
Der dritte (N-2,4 kb) wird durch die Rezeptoren für die Schilddrüsenhormone
(TR) und durch dessen onkogenes Homolog v-erbA wie auch durch den
Regulator NeP1 („negative
Protein 1”),
welches auch als „CTCF
(CCCTC-binding factor” (Burcin
et al., 1997; Darling et al., 1993; Kohne et al., 1993; Bhat et
al., 1994)) bezeichnet wird, erkannt. NeP1 bindet sich auf der Höhe des F1-Abschnitts
von 50 bp von N-2,4,
wohingegen der TR hauptsächlich
im Bereich des Abschnitts F2 von diesem Element bindet. Die homodimeren
oder heterodimeren Assoziationen mit den anderen nukleären Rezeptoren
(RAR, RXR) modulieren das Transkriptionsniveau von diesem Gen (Baniahmad
et al., 1990; Arnold et al., 1996). Der Phosphorylierungszustand
des TR-Rezeptors ist gleichfalls ein wichtiges Element. Schließlich spielt
der Faktor HNF-I alpha („Hepatic
nuclear Factor-I”),
der in den Zellen des Eileiters (Legedarms) exprimiert wird, eine
Rolle bei der Regulation des Lysozym-Gens. Der Promotor des Gens
enthält
zwei Regionen, die durch dieses HNF-I-Protein erkannt werden. Diese
Regulation der Expression des Gens des Lysozyms durch NHF-I scheint
auf der phylogenetischen Ebene zwischen den Vögeln und den Säugetieren
verloren gegangen zu sein (Grajer et al,. 1993). Das
Gen des Conalbumins, welches auch als Ovotransferrin bezeichnet
wird, wurde zuallererst beim Menschen identifiziert. Es erstreckt
sich über
mindestens 33,5 kbp und umfasst 17 Exons. Das Gen vom Huhn ist gleichfalls
in 17 Exons und 16 Introns organisiert, aber nur über 10,5
kb. Die mRNA hat eine Größe von 2376
bp (Cochet et al., 1979; Jeltsch und Chambon, 1982; Jeltsch et al.,
1987; Schaeffer et al., 1987). Es wurden sehr wenig Untersuchungen über die
Regulation der Expression von diesem Gen ausgeführt. Die Regulation der Stabilität der mRNA
ist besonders. Das Molekül,
das zu der Familie der Transferrine gehört, bindet Eisen in ionischer
Form und reguliert die Stabilität
und das Transkriptionsniveau von seiner eigenen mRNA. Auch wenn
viele Untersuchungen an dem humanen Transferrin und seinem Rezeptor
(CD71) ausgeführt
worden sind, gibt es wenig Dokumentation hinsichtlich der Vogel-Formen. Bei
den Eukaryoten ist die homologe Rekombination ein natürliches
Ereignis, das überdies
im Moment der Meiose auftritt, um in einer grob vereinfachenden
Aussage eine Durchmischung der Gene und der Allele sicherzustellen.
Die Rolle der homologen Rekombination ist während einer mitotischen Teilung
viel delikater zu interpretieren. Die homologen und nicht-homologen
Rekombinationsphänomene
scheinen gleichwohl entscheidend zu sein, sobald das genetische
Material geschädigt
ist. Zahlreiche Situationen im Leben einer Zelle und eines Organismus
exponieren so das genetische Material. Beispielsweise ein unerwarteter
Replikationsfehler, eine mit Strahlungen verbundene radikalische
Ionisation, ein oxidativer Stress, die Wirkung von Endonukleasen
oder von Topoisomerasen, die mit der mitotischen Segregation verbundene
mechanische Belastung, können
zu zufälligen
doppelsträngigen
Schnitten führen.
Wenn auch die Hefe die homologen Reparaturmechanismen favorisiert,
scheint die höhere
eukaryotische Zelle die nicht-homologe Zusammenfügung bzw. Reparatur von diesen
Schnitten zu bevorzugen, obgleich auch die homologe Reparatur beobachtet
wird. Aber in allen Fällen,
da die genetische Unversehrtheit eine Voraussetzung für eine jegliche
Teilung ist, setzt die Zelle ein ganzes Ensemble von Genen, die
damit beauftragt sind, diese Schnitte zu reparieren, in Gang, um
sich dieser für
das geschädigte
Material kritischen Situation zu stellen. Anfänglich in dem Hefesystem beschrieben, wurden
die Rad-Gene (rad51, rad52, rad53 und rad54) (Takata et al., 2000;
Morrison und Takeda, 2000; Bell et al., 1999; Shinohara et al.,
1997; Bezzubova et al., 1997; Ivanonv und Haber, 1997; Porter et
al., 1996), die an der Reparatur durch homologe Rekombination beteiligt
sind, auch in den höheren
Organismen identifiziert. Die Homologen Rad51 und Rad54 vom Huhn
sind kloniert worden. Die DNA und das Protein weisen starke Homologien
zu den Sequenzen aus der Hefe, der Maus und dem Menschen auf (Essers
et al., 1997; Dronkert et al., 2000). Es werden auch Ähnlichkeiten
bei der Wirkung beobachtet, aber auch Unterschiede, insbesondere
was die Wirkung von Rad51 angeht (Bezzubova et al., 1993). Ebenso
sind die humanen Homologen der Gene ku70 und ku8o von der Hefe zugleich
an den nicht-homologen Reparaturen und an den Rekombinationsphänomenen,
die bei den Umlagerungen der Genorte der Immunglobuline beobachtet
werden, beteiligt. Man beginnt, sich den Beziehungen zwischen diesen
verschiedenen Akteuren ebenfalls anzunähern, und hierbei wird die
Bildung von Proteinkomplexen nachgewiesen, deren Regulationen ihrerseits
komplex sind (Morrison et al., 2000; Morrison et al., 1999; Yamaguchi-Iwai
et al., 1999; Takata et al., 1998; Yamaguchi-Iwai et al., 1998).
So kann eine nicht erschöpfende
Liste von Proteinen, die die Rekombination vereinfachen (Rekombinase),
angegeben werden, von dem einfachen bakteriellen Protein RecA (Shcherbakova
et al., 2000) bis zu den Mitgliedern der Rad-Familie, aber auch
Proteine, wie BraC1 (Moynahan et al., 1999). In
einem Ansatz, der Ereignisse von homologer Rekombination vereinfachen
soll, wird die Wirkung von Rad 54 zum Zeitpunkt der Einführung des
homologen Rekombinationsvektors ausgewertet. So sind zwei Ansätze möglich. Der
erste besteht darin, eine Cotransfektion eines Expressionsvektors
von einem der Proteine der Rad-Familie, insbesondere Rad 54, mit
dem homologen Rekombinationsvektor vorzunehmen. Der zweite besteht
darin, die Zellen mit einem konditionellen Expressionsvektor von
einem der Proteine aus der Rad-Familie, insbesondere Rad 54, zu
transfizieren. In diesem Falle werden die Zellen zuallererst selektioniert, stabilisiert
und etabliert, um erneut mit einem homologen Rekombinationsvektor
transfiziert zu werden, dann induziert, um das Protein aus der Rad-Familie
maximal zu exprimieren. Die Wirkung von Rad54, die auf der Ebene
der Transfektionseffizienz getestet worden ist, ist in der nachfolgenden
Tabelle 4 aufgeführt: Um
die Bedeutung von einem der Proteine aus der Rad-Familie zu testen,
wird das Expressionsplasmid pRad54 (Expressionsplasmid des Proteins
RAD54 unter der Kontrolle des CMV-Promotors) in Gegenwart des Plasmids
pOvaRH, einem Rekombinationsplasmid, welches keine cDNA von Interesse
enthält,
für eine Cotransfektion
eingesetzt. Die gesamte Menge von Plasmid beträgt 5 μg von pOvaRH in Gegenwart von
variablen Mengen von pRad54. Da die molare Ergänzung durch ein „Carrier”-Plasmid
beitragen wird, hat das pMCS5 kommerzieller Herkunft ungefähr die gleiche
Größe wie pCMV
Rad54. Die Stammzellen S86N werden zu 1 × 106 Zellen
pro Schale in Proliferationsmedium in Gegenwart von durch Bestrahlung
inaktiviertem Feeder ausgesät.
Die Transfektionsmischung wird zugesetzt und die Selektion mit Neomycin
während
etwa 7 Tagen angewendet. Die Klone werden dann nach Fixierung mit
Methanol und Anfärbung
mit Wright/Giemsa gezählt. Als
Schlussfolgerung scheint das Vorhandensein von Rad54 die Effizienz
der Gewinnung von Klonen nicht wesentlich zu modifizieren. Einer
der Ansätze,
die gewählt
wurden, um die exogenen Proteine zu produzieren, besteht in deren Anhäufung im
Ei. Die im Rahmen der Erfindung eingesetzte substitutive Ersetzungsstrategie
nutzt die Verwendung eines aktiven Systems (aber gegebenenfalls
passiven Systems für
spezielle Proteine, die dazu die Fähigkeit haben) zur Anhäufung des
exogenen Proteins im Eiweiß aus.
So werden die kodierenden Phasen unter die Abhängigkeit des endogenen Promotors
des endogenen Zielgens gestellt und werden mit einem Referenz-Signalpeptid
fusioniert. Die Funktion des Signalpeptids besteht darin, die Translokation
des Proteins in Richtung der Außenseite
der Zelle durch die verschiedenen konstitutiven Elemente des sekretorischen
Apparats der Zellen (Retikulum, Golgi...) sicherzustellen. Die Signalpeptide
sind dazu bestimmt, im Verlauf des Reifungsprozesses des Proteins
abgespalten zu werden. Jenes häuft
sich dann in der physiologischen Flüssigkeit und insbesondere im
Eiweiß an.
Man setzt entweder die Signalpeptide der endogenen Proteine oder
Signalpeptide von anderen Molekülen,
bekannte Sequenzen, um korrekt behandelt zu werden, ein. Insbesondere wird
das Signalpeptid des Lysozyms präferentiell
ausgewählt,
denn seine Struktur ist gut bekannt und die 18 ersten Aminosäuren werden
während
der Exportierung des reifens Proteins in das Eiweiß abgespalten.
Außerdem
gehört
dieses Signalpeptid zu einem Protein, das sich von Natur aus im
Eiweiß anhäuft. Unter
den anderen eingesetzten Signalpeptiden findet man jene der Interleukine,
jene von bestimmten Membranrezeptoren, jene von durch verschiedene
Zellarten sekretierten Wachstumsfaktoren, jene von anderen Proteinen,
von denen bekannt ist, dass sie in verschiedenen biologischen Systemen
sekretiert werden. Die Liste ist nicht einschränkend. Das
kommerzielle Plasmid pCl Néo
(Promega, Madison, USA) wird durch HindIII-Verdau geöffnet und intramolekular
mit sich selbst ligiert, um das Plasmid pCI Néo (Δ HindIII) zu erhalten, das das
Gen der Neomycinphosphotransferase unter die direkte Abhängigkeit
des CMV-Promotors stellt. Die polyA-Ausgangssequenz wird bewahrt. Die
cDNAs von Interesse werden hergestellt durch PCR-Amplifizierung
mit Das
amplifizierte PCR-Fragment wird durch SmaI hydrolysiert (unter der
Voraussetzung der Abwesenheit von dieser Stelle in der cDNA) und
in die SmaI-Stelle in einem pMOS5-Vektor (Mobitech, Deutschland), in welchen
vorab eine polyA-Sequenz von 277 bp in die XbaI/XhoI-Stellen subkloniert
worden ist, kloniert. Diese polyA-Sequenz stammt ihrerseits aus
einer Hydrolyse des Vektors pIRESHygro (Clontech) durch XbaI/XhoI. Die
Orientierung des Fragments wird dann durch einfachen Verdau mit
der von der cDNA von Interesse gelieferten Karte kontrolliert. Im
Falle des Vorhandenseins einer SmaI-Restriktionsstelle in der cDNA
von Interesse wird das amplifizierte PCR-Fragment der Wirkung der
T4-DNA-Polymerase
unterworfen, um die Enden stumpf zu machen, und direkt in den an
der SmaI-Stelle
geöffneten
Vektor pMCS5 (stumpfes Ende) kloniert. Die
genomische DNA von 6 bis 9 Tage alten Embryos von verschiedenen
Stämmen,
darunter der Stamm CNR (Cou Nu Rouge) und der Stamm S86N, wird entweder
gemäß einer
klassischen Methode mit einem SDS-Proteinase K-Lysepuffer, gefolgt
von Extraktionen mit Phenol und Phenol-Chloroform, oder mit Hilfe eines
Präparationskits
(beispielsweise Promega- oder Qiagen-Kit) extrahiert. Die verschiedenen
Protokolle liefern Ausbeuten und Qualitäten von DNA, die mit deren
jeweiliger Verwendung bei der Herstellung einer genomischen DNA-Bank
und einer Gewinnung von PCR-Fragmenten durch direkte Amplifizierung
verträglich
sind. In
dem ersten Falle wird die genomische DNA partiell durch das Enzym
Sau3Al verdaut und die erzeugten Enden durch die Klenow-Polymerase
in Gegenwart von zwei der vier Nukleotide, die für die Synthese notwendig sind,
aufgefüllt.
Daraus resultiert eine partielle Auffüllung, um die Bildung von konkatemeren
Inserts zu verhindern. Die so partiell aufgefüllten Inserts werden durch
Ligation in den Vektor λ-GEM-12
inseriert. Die aus dieser Vorgehensweise resultierenden Vektoren
werden in Kapside von Phagen verpackt. Als Beispiel wurden 3 unabhängige Isolate
erzeugt und ihr Titer bestimmt, wobei jedes Isolat auf etwa 3 × 106 pfu/ml titriert wurde. Die Qualität der Bank
wurde durch die Untersuchung von einigen zufällig ausgewählten inserts verifiziert.
Mit einer Ovalbumin-Sonde, erhalten durch genomische PCR mit ausgehend
von der veröffentlichten
Sequenz ausgewählten
Oligonukleotiden (Tabelle 3), erlaubt das Screening der Phagen,
Inserts zu erhalten, die in pBS SK+-Plasmidvektoren subkloniert werden,
um das Plasmid #72 zu erhalten. Diese inserts, die einer Restriktionskartierung
(darunter EcoRI und BamHI) unterworfen worden sind, wurden dann
sequenziert. Ein Vergleich zwischen dem veröffentlichten Gen (Ref. J00895
und Tabelle Nr. 3) und den beiden 5'- und 3'-Sequenzabschnitten des Vektors preRH
wie auch den Fragmenten des Klons #72 zeigt, dass die Homologien
auf der Nukleotidebene extrem hoch sind. Allein zwei Segmente, die
sich im Bereich der Introns 6 und 7 befinden, weisen geringfügige Abweichungen,
die über
einige zehn Nukleotide verteilt sind, auf (Ergebnis nicht gezeigt). Dieser
Vergleich zeigt die starke Konservierung von diesem Gen zwischen
den analysierten Stämmen. In
dem zweiten Fall werden verschiedene Oligonukleotide, die ausgehend
von der veröffentlichten
Sequenz erhalten worden sind (Tabelle 3 oben) eingesetzt, um durch
genomische PCR die Fragmente, die die 5'- und 3'-Sequenzabschnitte (-Arme) der Vektoren
ergeben, zu erhalten. Für den 5'-Sequenzabschnitt
des Vektors pOvaRH amplifizieren die Oligonukleotide OvaL 2995S
und OvaL 80 AS, die bei –2995
bp bzw. bei +80 bp bezogen auf das ATG des Ovalbumingens gelegen
sind, eine Region von 3075 bp, die, wenn sie einer Hydrolyse durch
NcoI unterworfen wird, ein Fragment von 2860 bp freisetzt. Parallel
dazu wird ausgehend von der Sequenz, die das Signalpeptid des Lysozyms
spezifiziert, ein als lyso 1–18 – intron
bezeichnetes Fragment durch PCR-Amplifizierung an dem Genort des
Lysozyms mit den Oligonukleotiden Lyso 1–18 und GE-IN-AS hergestellt.
Dieses letztere Oligonukleotid enthält die 30 bp des künstlichen
Introns des Plasmids pCkNéo.
Die beiden Fragmente (Ova5' und
Lyso1-18 intron) werden unabhängig
durch NcoI hydrolysiert, gereinigt, dann zusammen ligiert und die
Amplifizierung an dem Ligationsprodukt mit den Oligonukleotiden
OvaL 2995S und GE-IN-AS liefert das Fragment Ova5' Lyso 1–18 von
3050 bp. Dieses PCR-Produkt wird mit EcoRI-Adaptoren ligiert, die
erlauben, diese Gesamtheit entweder an der EcoRI-Stelle in den Vektor
pBSK oder in den durch EcoRV zu stumpfen Enden verdauten Vektor
pMCS5 zu klonieren. Eine
letzte Hydrolyse von diesem Fragment Ova5'Lyso1-18 durch Bst981 und AscI setzt
das geschnittene Plasmid und ein kleines Fragment von 220 bp strangaufwärts von
dem ova5'-Fragment
frei. Diese kleine Deletion erlaubt, zwei Oligonukleotide Ova5'.1S und Ova5'.2S zu gestalten,
die später
für das
Screening der Rekombinationsereignisse eingesetzt werden. Das
geschnittene Plasmid wird der Wirkung der Klenow-Polymerase unterworfen,
um die Enden stumpf zu machen, bevor es mit AscI-Adaptoren ligiert
und intramolekular ligiert wird. Daraus resultiert ein Plasmid pOvaLyso,
das strangaufwärts
von dem Ova5'-Arm
eine einmalig vorkommende Stelle, die eine Linearisierung des endgültigen Vektors
erlauben wird, enthält. Für den 3'-Arm des Vektors
pOvaRH liefert die Amplifizierung der strangabwärts zwischen den Positionen
+1657 und –7707
bezogen auf das ATG des Ovalbumingens gelegenen Region mit den Oligonukleotiden OvaL
1657S und OvaL7707 AS ein Fragment von einer Größe von 6050 bp. Dieses Fragment
wurde in die EcoRV-Stelle von pMCS5 subkloniert, um das Plasmid
pMCS5-Ova3' Sinn
und pMCS5-Ova3' Antisinn
zu erzeugen. Ausgehend von dem Plasmid pMCS5-Ova3' Antisinn erlaubt
ein Verdau durch HpaI (stumpfe Enden) und NarI (5'-überhängend), das Fragment von 6
kb zu erhalten, das in pBS-SK an der ApaI- (mit stumpfen Enden versehen)
und AccI-Stelle (kompatibel mit NarI) subkloniert wird, um das Plasmid
pBS-SK Ova3' zu
erhalten. Für den 5'-Sequenzabschnitt
des Vektors pLyso RH haben die proximalen Amplifizierungen mit den Oligonukleotiden
lyso 1789 S und GE-IN-Ceu-AS erlaubt, ein Fragment zu isolieren
und dieses direkt nach Behandlung mit T4-DNA-Polymerase und Phosphorylierung
durch die Polynukleotidkinase in den kommerziellen Vektor pMCS5,
der mit stumpfen Enden an der EcoRV-Stelle geöffnet worden ist, subzuklonieren.
Dieses Fragment pLyso 5' enthält die teilweise
nicht veröffentlichte
2900 bp lange Sequenz des proximalen Abschnitts des Lysozym-Gens
wie auch das Äquivalent
der 18 ersten Aminosäuren
des Exons 1, welches dem Signalpeptid des Lysozyms entspricht. Dieses
Fragment enthält
folglich ein Spleiß-Donorstelle wie auch
die Sequenz des kleinen künstlichen
Introns, einer durch das Oligonukleotid AS hinzugefügten Sequenz,
wie zuvor hinsichtlich des 5'-Sequenzabschnitts
(-Arms) des Vektors
pova5' beschrieben. Für den 3'-Sequenzabschnitt
des Vektors pLysoRH haben die distalen Amplifizierungen mit den
Oligonukleotiden Lyso 2859S und Lyso 3185 AS erlaubt, ein Fragment
von 2150 bp 3' von
dem Gen zu isolieren. Diese Oligonukleotide tragen an ihrem Ende
Scel- und MluI-Stellen. Dieses einer Behandlung mit der T4-DNA-Polymerase,
einer Phosphorylierung durch die Polynukleotidkinase unterworfene
und direkt in das durch EcoRV mit stumpfen Enden geöffnete Plasmid
pMCS5 ligierte Fragment liefert das Plasmid plyso3'. Die Hydrolyse von
diesem Plasmid durch AflII/AvrII, gefolgt von der Wirkung der T4-DNA-Polymerase
und einer intramolekularen Ligierung erlaubt, das Plasmid pLyso3' zu erzeugen. Der
endgültige
Zusammenbau des RH-Vektors durch aufeinanderfolgende Hinzufügungen von
verschiedenen Kassetten (Selektionskassette, Insertion der cDNA
oder des Gens von Interesse...) erfolgt gemäß verschiedenen Schematas.
Es wird ein Schema beschrieben, ohne dass dieses andere Möglichkeiten,
die in Abhängigkeit
von den in bestimmten cDNAs von Interesse vorhandenen Restriktionsstellen
existieren, ausschließen
soll. Das Plasmid pCMV NéoOva3' wird durch Ligation
des Fragments Ova3',
das aus der Reinigung nach Schnitt des Plasmids pOva3' durch BamHI resultiert,
mit dem gereinigten und dephosphorylierten Fragment pCMV Néo polyA,
erhalten durch BgIII/BamHI-Hydrolyse des Plasmids pCINeo(Δ-hindIII),
konstruiert. Es werden gleichfalls einige Modifizierungen des strangabwärts von
dem Ova5'lyso-Insert
vorhandenen Polylinkers vorgenommen, bevor die beiden Fragmente
Ova5'lyso und pCMV-Néo-Ova3' in einem Vektor
pOvaRH zusammengefügt
werden ( – die einmalig
vorkommende AscI-Stelle, die die Linearisierung des Vektors vor
Transfektion erlaubt, Der
Vektor pOva RH ist dann einsetzbar, um die verschiedenen cDNAs von
Interesse zu inserieren. Das
Fragment Lyso 3',
erhalten durch die Hydrolyse des Plasmids pLyso3' durch Mlul, wird in das Plasmid plyso5', das zuvor geöffnet und
nach Hydrolyse durch Mlul dephosphoryliert worden ist, ligiert.
Daraus resultiert das Plasmid pLyso5'Lyso3', in welches das gereinigte und dephosphorylierte
Fragment pCMV Néo
polyA, erhalten durch NotI/MluI-Hydrolyse
des intermediären
Plasmids pMCS5 CMVNéoPolyA.
Diese letztere Subklonierung erfolgt, um die Selektionskassette
5' mit der NotI-Stelle
zu flankieren, um diese Stelle für
die Klonierungen der cDNAs von Interesse zu bewahren. Sie wird durch
Insertion des BgIII/BamHI-Fragments des Plasmids pCINéo(Δ-hindIII)
in das an der EcoRV-Stelle geöffnete
und dephosphorylierte Plasmid pMCS5 erhalten. Der
erhaltene Vektor pLysoRH enthält
aufeinanderfolgend von 5' nach
3': Der
Vektor plysoRH ist dann einsetzbar, um die verschiedenen cDNAs von
Interesse zu inserieren ( Die
cDNAs von verschiedenen Proteinen von Interesse oder Modellproteinen
werden in diese Vektoren pOvaRH und pLyso RH inseriert. Nicht erschöpfend besteht
die Strategie darin, die cDNA gemäß dem zuvor präsentierten
Schema herzustellen und diese in den an der NotI-Stelle geöffneten
Vektor zu inserieren. Varianten hinsichtlich der Reihenfolge der
Klonierungen der verschiedenen Abschnitte können erforderlich sein. Es
erfolgen partielle Sequenzierungen oder Sequenzierungen zur Gänze, um
die korrekte Aneinanderreihung der verschiedenen inserierten Elemente
zu verifizieren. So wurden als nicht erschöpfende Beispiele Vektoren mit
den cDNAs des Erythropoietins pOvaRH Epo, pLysoRH Epo, der beta-Galactosidase pOva
RH LacZ, pLysoRH LacZ, des Heliomycins (antibakterielles Peptid
pO-vaRH Hélio, pLysoRH
Hélio)
konstruiert. Die Ein
besonderer Fall ist die Anwendung der Vektoren pOvaRH und pLyso
RH auf die Herstellung von Molekülen,
die ihr eigenes Signalpeptid aufweisen. Die eingesetzte Konstruktionsstrategie
unterscheidet sich ein wenig von jenen, die zuvor beschrieben worden
sind. Beispielsweise ist die cDNA des Interleukins 11 ein Nukleotidfragment
von 1100 bp, das in die EcoRI-Stellen in den Vektor pBS SK subkloniert
ist. Wenn es folglich nicht notwendig ist, die cDNA von Interesse
mit dem Signalpeptid des Lysozyms zu fusionieren, wird die cDNA direkt
im Raster auf der Höhe
des ATG des Ovalbumins inseriert, was zu einer Fusion auf der Höhe der zweiten Aminosäure des
Interleukins 11 führt.
In diesem Beispiel wird die cDNA von IL-11 der Wirkung der Taq-Polymerase
in einer PCR-Reaktion in Gegenwart von zwei Sinn- und Antisinn-Oligonukleotiden,
die die HindIII- und XbaI-Stellen aufweisen, unterworfen. Dieses
Fragment wird so der doppelten HindIII/XbaI-Hydrolyse unterworfen
und in den Vektor pMCS5, der seinerseits geöffnet an den HindIII/XbaI-Stellen
vorbereitet worden ist nach doppelter Hydrolyse durch diese Enzyme
und Dephosphorylierungsbehandlung mit CIAP, ligiert. Aus dieser gerichteten
Klonierung resultiert das Plasmid pMCS5-II-11. Dieses Letztere wird dem HpaI/NsiI-Verdau
unterworfen, das Fragment wird gereinigt und in das Plasmid pOva5' AS, das seinerseits
an der NcoI/PstI-Stelle geöffnet
und zu stumpfen Enden durch die Wirkung der Klenow-Polymerase repariert
worden ist, ligiert. Daraus resultiert das Plasmid p0va5' IL-11. Dieses
Plasmid p0va5'-II11
wird dem Verdau durch NotI unterworfen, das Insert wird gereinigt
und mit dem Plasmid pCMVNéoOva3', das seinerseits
durch Not' hydrolysiert
und dephosphoryliert worden ist, ligiert. Daraus resultiert das
Plasmid pOvaRH II-11, das gebildet wird aus: Eine
analoge Strategie erlaubt, den Vektor pLysoRH IL-11 in Gegenwart
des endogenen Signalpeptids von Lysozym und durch Delektion des
Signalpeptids von IL-11 und umgekehrt durch Deletion des endogenen Signalpeptids
von Lysozym und Hinzufügen
des Signalpeptids von IL-11 zu erhalten. Es
werden zwei allgemeine Transfektionsmethoden eingesetzt: die Elektroporation
und die Transfektion durch Liposome. Es scheint, dass in dem Mäuse-System
im Allgemeinen die Elektroporation gewählt wird, um die Rekombinationseffizienz
der homologen Rekombinationsvektoren zu erhöhen. Gleichwohl sind wenig vergleichende
Untersuchungen ausgeführt
worden. Die
Elektroporation ist ein Verfahren, welches erlaubt, eine DNA von
sehr variabler Größe in eine
Zelle eintreten zu lassen, indem durch einen elektrischen Schlag
eine vorübergehende
Pore auf der Ebene der Zellmembran erzeugt wird. Unter den verschiedenen
Parametern, die die Wirksamkeit dieses Experiments beeinflussen
können,
kann man die Zellkonzentration (von 1 bis 10 × 106 Zellen),
die das Signal leitende Lösung, welche
die Mischung von Zellen und DNA enthält (PBS, serumfreies Medium...),
die Konzentration von im Allgemeinen linearisierter DNA (von 5 bis
25 μg),
den Abstand der Elektroden (Küvetten
oder einfache Elektroden direkt in der Kulturschale oder der Kulturvertiefung),
die Amplitude des Signals, welche von der angewandten Kapazitanz
(von 10 bis 1000 μF)
und der angelegten Spannung (von 5 bis 100 mV für Rechtecksignale und von 200
bis 350 V für
die Nicht-Rechtecksignale),
die Anzahl von abgegebenen elektrischen Schlägen und die Form von diesem
bzw. diesen angewandten elektrischen Schlag bzw. Schlägen aufführen. Unter
Form versteht man die Art und Weise, in der ein und dieselbe elektrische
Ladung abgegeben wird, insbesondere gemäß einem Rechtecksignal, Nicht-Rechtecksignal,
exponentiellen inversen Signal... Die
Tabelle 5 veranschaulicht einige von diesen Parametern: Die
Elektroporation des linearen Plasmids pClNéo erfolgt mit Hilfe eines
BioRad-Elektroporationsgeräts (Gene
Pulser II). Die dissoziierten, mit einem serumfreien Medium gewaschenen
und auf eine Konzentration von 5 × 106 Zellen
in 0,8 ml eingestellten Zellen werden in eine Elektroporationsküvette (4
mm-Kammer) gegeben. 5 bis 20 μg
linearisiertes Plasmid, hergestellt in TE, werden zu den Zellen
hinzugegeben und man lässt
die Mischung 5–10
min bei Umgebungstemperatur stehen, dann wird der elektrische Schlag
mit einer auf 560 μF
festgelegten Kapazitanz angewendet. Die Zellen werden dann etwa
10 min bei 4°C
platziert, dann in Kulturschalen mit Feeder unter den Kulturbedingungen
von diesen Zellen ausgesät.
Klassischerweise wird das Äquivalent
von einer Küvette,
d. h. 5 × 106 Zellen, in zwei 100 mm-Kulturschalen verteilt. Die
Tabelle führt
3 mit 15 μg
Plasmid bei 270 V ausgeführte
Reihen auf. Die Klone werden nach Selektion mit Neomycin während 7
Tagen (250 μg/ml,
Fixierung mit Methanol und Anfärbung
mit Wright/Giemsa gezählt. Unter
Liposomen versteht man ein jegliches chemisches Molekül, welches
unter anderem Merkmale von kationischen Lipiden aufweist, allein
oder assoziiert mit anderen Elementen. Die Wirksamkeit von verschiedenen
Liposomen wurde an verschiedenen Zellarten verglichen. Da die embryonalen
Stammzellen fragil sind, ist das Vorhandensein von Serum während der
Transfektion ein wichtiger Faktor. Es wurden verschiedene kommerzielle
Materialien getestet. Daraus geht hervor, dass die Wirksamkeit und
die Toxizität
von diesen Komponenten variabel sind (Pain et al., 1999). Außerdem erlaubt
das Vorhandensein von Serum, die relativen toxischen Wirkungen der
Exposition der Zellen gegenüber
den Lipofektionsmaterialien zu verringern und so die Chancen zu
bewahren, rekombinante Zellen in einem zufrieden stellenden physiologischen
und morphologischen Zustand zu erhalten. Ein Vorteil der Verwendung
der Liposome ist unter anderem die geringe Anzahl von Zellen, die
pro Versuch eingesetzt werden. Die nachfolgende Tabelle 6 veranschaulicht
eine relative Transfektionseffizienz mit einem einfachen Expressionsvektor: Die
Zellen werden in ihr jeweiliges Medium in einer Dichte von 5 × 105 bis 106 Zellen
pro 100 mm-Schale für
jede Zellart ausgesät.
Die Transfektionsmischung, welche von 10,5 μg Liposome/3,5 μg DNA bis
15 μg Liposome/5 μg DNA pro
100 mm-Schale variieren kann, wird je nach Fall 3 bis 15 h auf die
Zellen gegeben. Die Selektion mit Neomycin wird mit einer Konzentration
von 250 μg/ml
angewendet und die Klone erscheinen 5 bis 10 Tage nach der Transfektion.
Die Klone werden entnommen und durch PCR analysiert, um die Rekombinationsereignisse
nachzuweisen. Ein
typisches Amplifizierungsbeispiel besteht in einer Klonierung in
Vertiefungen einer Platte mit 24 Vertiefungen, in einer Passage
in Vertiefungen einer Platte mit 12 Vertiefungen, dann einer Amplifizierung
in einer 60 mm- oder 100 mm-Schale. Die auf optimale Weise zwischen
der Passage von den Vertiefungen einer Platte mit 24 Vertiefungen
zu den Vertiefungen einer Platte mit 12 Vertiefungen ausgeführten Screeningschritte erlauben,
die Amplifizierung allein auf die Klone, die durch PCR als positiv
nachgewiesen worden sind, zu beschränken. Pro Vertiefung einer
Platte mit 6 Vertiefungen erhält
man im Allgemeinen 5 × 105 bis 106 Zellen.
So werden als lebensfähig
lediglich die durch PCR als positiv gescreenten Klone eingefroren.
Die Amplifizierung für
eine Southern-Blot-Analyse betrifft gleichfalls lediglich eine geringe
Anzahl von Klonen. Die
Tabelle fasst verschiedene Beispiele, die mit Zellen unterschiedlicher
Natur und mit unterschiedlichen Passagen erhalten worden sind, zusammen.
Es ist notwendig, festzustellen, dass die Transfektionseffizienzen
und die Rekombinationseffizienz je nach den Vektoren variabel scheinen
und für
ein und denselben Vektor von der Physiologie (Alter, Zustand...)
der eingesetzten Zellen abhängig
sein können. Die
Effizienz hinsichtlich stabilen Transfektionen wird anhand der Anzahl
von Klonen, die nach Selektion durch das Arzneimittel, das durch
das in der Selektionskassette vorhandene Gen entgiftet wird, erhalten werden,
abgeschätzt.
In den berichteten Fällen
wird im Allgemeinen Neomycin eingesetzt. Abhängig von dem Vorhandensein
eines unterschiedlichen Resistenzgens in dem für die Transfektion eingesetzten
Vektor können andere
Arzneimittel bzw. Wirkstoffe eingesetzt werden. Unter den klassischen
Arzneimitteln findet man Neomycin, Hygromycin, Puromycin, Phleomycin,
Zeomycin, Blasticidin, Viomycin... Es können andere Selektionsmedien
eingesetzt werden, wie das Hinzufügen von Basenanaloga, die klassischen
Selektionsmedien, wie das HAT-Medium.... Die
Klone werden am Ende des Selektionszeitraums, welches dem Verschwinden
der Zellen in den Vergleichsschalen des Experiments entspricht,
gezählt.
Diese Vergleichsproben sind entweder nicht transfizierte Zellen
oder mit einem Vektor, der kein Resistenzgen für das eingesetzte Antibiotikum
enthält,
transfizierte Zellen. Die
embryonalen Fibroblasten vom Huhn („Chicken Embryonic Fibroblasts”) werden
ausgehend von einem Inkulturnehmen von 9 bis 13 Tage bebrüteten Hühnerembryos
gemäß einem
gut etablierten und den Fachleuten auf diesem Gebiet bekannten Protokoll
erhalten. Diese primären
Zellen werden amplifiziert und während
einer wenig bedeutenden Anzahl von Passagen (im Allgemeinen unter
30) in Kultur gehalten, denn die Zellen zeigen schnell (im Allgemeinen
ab den Passagen 15 bis 20) eine progressive Phase von Anhalten der
Proliferation und einen Eintritt in die Seneszenz, der durch eine
geringere Teilungsrate, eine immer weniger fibroblastische zur Schau
gestellte Morphologie gekennzeichnet ist. Diese Seneszenzphase führt zu dem
Verschwinden der Zellen bis auf sehr seltene Fällte, wo die spontane Etablierung
von fibroblastischen Vogel-Zellen und insbesondere von Hühnerzellen
beobachtet worden ist (Linie DF-1) (Himly et al., 1998; Kim et al., 2001). Die
Transfektionen durch die homologen Rekombinationsvektoren erfolgen
folglich während
früher Passagen
(von 1 bis 10), um zugleich die Möglichkeit, die gegenüber der
Selektion resistenten Klone zu selektionieren und zu amplifizieren,
zu optimieren, aber auch um ein etwaiges karyotypisches Derivat
und den schnellen Eintritt in die Seneszenz zu begrenzen. Im Allgemeinen
sind die erhaltenen Klone isolierbar, aber nicht oder wenig amplifizierbar.
Im Gegenzug können
sie als potentielle Quellen für
modifizierte Kerne für
nukleare Klonierungsexperimente angesehen werden. Die
Zelllinie LMH ist eine Hepatozytenlinie vom Huhn, erhalten durch
chemische Kanerzogenese (Kawaguchi et al., 1987). Diese Linie ist
auf karyotypischer Ebene charakterisiert worden und weist eine bedeutende
Polyploidie auf. Die Zellen werden im Allgemeinen in DMEM oder HamF12
in Gegenwart von 8% fötalem Kälberserum,
2% Hühnerserum,
10% TPB und von Antibiotika gehalten. Diese Zellen weisen einen
interessanten dendritischen Zelltyp, der von den Monozytenzellen,
Fibroblastenzellen und Stammzellen sehr verschieden ist, auf und
weisen eine relative Empfindlichkeit gegenüber verschiedenen Steroidhormonen auf.
Die Transfektionen von insbesondere diesen Zellen mit pOvaRH-Rekombinationsvektoren
haben erlaubt, Klone und Rekombinationsereignisse zu erhalten. Die
Tabelle 6 veranschaulicht die Ergebnisse von diesen Transfektionen. 6.3.3
Linie HD11 (Vergleichsbeispiel) Die Zelllinie HD11 ist eine Monozytenlinie
vom Huhn, die durch das Vorhandensein des Onkogens v-myc, das durch
ein Vogel-Retrovirus hinzugefügt
worden ist, unsterblich gemacht worden ist (Beug et al., 1979).
Diese Linie ist Produzent von Viren, die das Onkogen propagieren
können.
Bestimmte Klone könnten
gegebenenfalls keine Produzenten sein. Die Zellen werden im Allgemeinen
in HamF12-Medium, ergänzt
mit 5% fötalem
Kälberserum,
2% Hühnerserum,
10% TPB, gehalten. Wie in der Einleitung präzisiert, produzieren die aktivierten
Makrophagen Mengen von endogenem Lysozym. Indem sie durch die pLysoRH-Rekombinationsvektoren
modifiziert werden, können
diese Zellen folglich als Produktionsmodell für das in diesen Vektor inserierte
Molekül
dienen. Man realisiert so eine „knock in”-Produktionsstrategie. Die Tabelle 6
veranschaulicht vorstehend die Ergebnisse von diesen Transfektionen. Die
embryonalen Stammzellen vom Huhn, die in den Beispielen eingesetzt
werden, sind Stammzellen, die in vivo durch das Inkulturnehmen von
Blastodermen vom Huhn erhalten worden sind (Pain et al., 1996; Die
Tabelle 6 oben und die Tabelle 7 unten veranschaulichen die Ergebnisse,
die mit verschiedenen Vektoren erhalten worden sind. Es erweist
sich, dass diese Stammzellen an verschiedenen Genorten durch einen
Ansatz von homologer Rekombination modifizierbar sind. 0,8 × 106 S86N16-Zellen in verschiedenen Passagen
werden mit 5 μg
pCINéo-Vektor
mit 15 μg Fugène 6 (Roche)
in seiner zirkulären
Form transfiziert. Nach Anwendung der Selektion mit Neomycin durch Zugabe
von G418 (250 μg/ml)
während
5 bis 8 Tagen werden Klone erhalten. Die Schalen werden mit Methanol
(100%) fixiert und eine Anfärbung
mit Giemsa erlaubt, die Klone schnell zu zählen. Dieses Beispiel zeigt
in unabhängigen
Versuchen, dass die Zellen stabil mit einfachen Vektoren transfizierbar
und selektionerbar sind und dass die Klone amplifiziert werden können. Gleichwohl
sind die Niveaus der Expression der Transgene von einem Klon zum
anderen sehr variabel, wie man dies leicht sichtbar machen kann,
indem beispielsweise die Fluoreszenzintensität des Markers E-GFP ausgewertet
wird. Unter
physiologischem Zustand versteht man den Differenzierungs-, Transkriptions- und Aktivierungszustand
der eingesetzten Zellen. Da sie im Bereich der durch die einfachen
Vektoren und die homologen Rekombinationsvektoren zielgerichtet
angesteuerten Genorte modifiziert sind, zeigen die Zellen eine Reaktion auf
physiologischer Ebene. Es erweist sich folglich als nützlich,
diese abhängig
von ihrer zukünftigen
Verwendung zu verfolgen, von denen die hauptsächlichen die folgenden sind: Die
makroskopisch und mikroskopisch beobachteten Klone werden entnommen
und direkt in eine Platte mit 24 Vertiefungen gegeben. Es ist wichtig,
die Klone so früh
wie möglich
zu entnehmen, um die Mischungseffekte zwischen Klonen zu begrenzen.
Die Herkunft der Klone gemäß den verschiedenen
Schalen wird erwähnt,
um beispielsweise das Unabhängigkeitsniveau
von zwei positiven Klonen zu kontrollieren. Die Proliferationsbedingungen
der Klone sind ungefähr
die gleichen wie jene, die für
die Ausgangszellen eingesetzt wurden. Die
Klone werden, einmal entnommen, täglich beobachtet und sobald
die Dichte der Zellen dies erlaubt, werden die Klone dissoziiert
und für
eine Analyse amplifiziert. Die
Zellen der in Proliferation befindlichen Klone in den Vertiefungen
werden gewaschen, dissoziiert und vorbereitet, um einerseits für eine Amplifizierung
erneut ausgesät
zu werden und andererseits um analysiert zu werden. Die DNA wird
prinzipiell mit Hilfe von klassischen Techniken, darunter kommerzielle
Kits, extrahiert. Es werden verschiedene Nachweise vorgenommen,
um die Rekombinationsereignisse zu screenen. Die Entwicklung von
PCR-Bedingungen erfolgt für
jedes Paar von Oligonukleotiden mit aus transfizierten, selektionierten
und nicht individuell klonierten Zellen extrahierter DNA. Dieser „Pool”-Ansatz
erlaubt, schnell zu testendes Material zu erhalten. Allgemein wird
eine erste PCR ausgeführt,
um den zielgerichtet angesteuerten endogenen Genort nachzuweisen,
um sich der Qualität
der Extraktionen und der DNA zu versichern. Es wird eine zweite
PCR ausgeführt,
um ein Rekombinationsereignis zwischen einem bezüglich des Konstrukts externen
Oligonukleotid und einer allen Vektoren aus ein und derselben Familie
gemeinsamen Sequenz, beispielsweise der Signalpeptidsequenz lyso
1–18,
nachzuweisen. Diese PCR wird als unterscheidend für die Rekombinationsereignisse
angesehen. An den positiven Klonen in dieser letzteren PCR werden
neue Reihen von PCR stets mit dem gleichen externen Oligonukleotid,
aber mit einem anderen, für
jede cDNA spezifischen Oligonukleotid derart ausgeführt, dass
man sich des Vorhandenseins und der allgemeinen fehlenden Modifizierung
der inserierten cDNA versichert. Diese PCR wird als unterscheidend
angesehen und erlaubt, die Beobachtung der positiven Klone zu verstärken. Die Als
Beispiel wird die DNA mit dem Promega-Kit (Madison, WS) extrahiert.
Der zelluläre
Bodensatz von jedem Klon wird in 300 μl Lysepuffer (Nuclei Lysis Solution,
NLS) in Gegenwart von 20 μg/ml
RNAse wieder aufgenommen. Das Lysat wird homogenisiert, 30 min bei
37°C inkubiert
und es werden 100 μl
Proteinpräzipitationspuffer
(Protein Precipitation solution, PPS) zugesetzt. Das Ganze wird
nach kräftigem
Mischen bei 4°C inkubiert.
Der Überstand
wird nach Zentrifugation abgenommen und 400 μl Isopropanol werden zugesetzt. Nach
Präzipitation
und Zentrifugation wird der Bodensatz in 70°c Ethanol gewaschen, getrocknet
und in 50 μl TE
(10.1) wieder aufgenommen. Die Volumen können auf proportionale Weise
für Extraktionen
mit mehr Zellen eingesetzt werden. Die erhaltene DNA-Menge wird
durch spektrophotometrische Quantifizierung abgeschätzt. Die
Extraktion eines zu einer Vertiefung einer Platte mit 24 Vertiefungen äquivalenten
Bodensatzes liefert etwa 0,5 bis 2 μg DNA je nach der Dichte der
Zellen in der Vertiefung. Die PCRs werden an etwa 100 bis 500 ng ausgeführt. Eine
andere PCR wird ausgeführt,
um durch Ausschluss das Geschlecht der positiven Klone mit für eine wiederholte
Region des Chromosoms W spezifischen Oligonukleotiden zu bestimmen.
Nur die weiblichen Klone (ZW) werden nachgewiesen werden können, die
männlichen
(ZZ) werden dies nicht. Die Die
DNAs von verschiedenen Klonen, die anhand von PCR hinsichtlich eines
homologen Rekombinationsereignisses als positiv nachgewiesen worden
sind, werden einer genomischen PCR unterworfen, um das Geschlecht
der Klone zu bestimmen. Die eingesetzten Oligonukleotide sind spezifisch
für das
Chromosom W des Huhns. Sie können
lediglich weibliche Klone nachweisen. In der Figur sind die Klone
hauptsächlich
männlich,
wie die eingesetzten Zellen (S85N16) mit der Ausnahme des Klons
A5-1, der von frühen
Zellen einer anderen Herkunft stammt (Zellen S86N 48). Um
die Ergebnisse zu verifizieren, die im Rahmen einer genomischen
PCR an den positiven Klonen mit den beiden hinsichtlich des Nachweises
eines Rekombinationsereignisses unterscheidenden PCRs erhalten worden
sind, wird eine Southern-Blot-Analyse mit aus den Klonen extrahierter
DNA ausgeführt.
Diese Analyse erlaubt zu verifizieren, dass: Die
DNA der Klone wird gemäß einer
klassischen Methode mit Proteinase K-SDS mit phenolischer Extraktion
oder mit Hilfe eines kommerziellen Extraktionskits (Qiagen, Promega)
extrahiert. In einem klassischen Ansatz der Molekularbiologie wird
eine variable DNA-Menge
der Hydrolyse durch verschiedene Enzyme unterworfen, um zu erlauben,
die Fragmente ausfindig zu machen, deren Größe abhängig von dem Rekombinationsereignis
unterschiedlich ist. Die
Tabelle 8 veranschaulicht den Nachweis der Rekombinationsereignisse
durch Southern-Blot abhängig
von den verschiedenen eingesetzten Sonden. Die
hydrolysierten DNAs werden einer elektrophoretischen Wanderung,
die die Fragmente nach ihrer Größe auftrennt,
unterworfen. Es wird das klassische Southern-Blot-Verfahren angewendet
mit einem Transfer auf eine Hybond N+-Membran (Amersham Pharmacia
biotech, Vereinigtes Königreich).
Die für
die Hybridisierung eingesetzten Sonden werden entweder durch PCR
oder durch Reinigung eines Plasmidfragments, das die verschiedenen
Gene von Interesse enthält,
erhalten. Als Beispiel werden die Sonden lacZ (1159 bp) und Néo (354
bp) durch PCR ausgehend von Plasmid-Matrix erhalten. Die Sonde Ovalbumin
3' ist eine Intron-Sonde
von 800 bp, erhalten ausgehend von genomischer DNA. Die Sonde Ovalbumin
5' ist eine Intron-Sonde
von 870 bp. Diese Sonden werden durch PCR erhalten. Die
gereinigten Sonden werden mit alkalischer Phosphatase mit Hilfe
des Kits AlkPhos (Amersham Pharmacia Biotech, Vereinigtes Königreich)
markiert und können
mehrere Monate bei –20°C aufbewahrt
werden. Für
den Nachweis werden die Membranen unter Bewegung in einer Menge
von 1 ml/cm2 in einem Hybridisierungspuffer
vorhybridisiert und dann bei 55°C
durch Zugabe der markierten Sonde (5 ng/ml) hybridisiert. Die Membranen
werden dann bei 65°C
in dem primären
Puffer, der 2 M Harnstoff, 150 mM NaCl, 50 mM NaPhosphat, 1 mM MgCl2, 0,1% SDS und 0,2% „blocking agent”, das durch
den Kit bereitgestellt wird, enthält, dann in dem sekundären Puffer,
welcher 50 mM Tris-HCl, pH 10,0, 100 mM NaCl enthält, bei
Umgebungstemperatur gewaschen. Das
einsatzfertige Sichtbarmachungssystem durch Chemolumineszenz wird
zu der gewaschenen Membran hinzugesetzt (1 ml/cm2)
und die Intensität
der Banden wird durch Autoradiographie sichtbar gemacht. Die Größe der nachgewiesenen
Banden wird mit der erwarteten Größe verglichen. Das
Kriterium, um die Klone beizubehalten, ist eine gute Entsprechung
zwischen der erwarteten Größe und jener,
die beobachtet wird. Es wurden ergänzende Hybridisierungen ausgeführt (Ergebnis
nicht gezeigt). Die Die
verschiedenen Kriterien, die eingesetzt wurden, um die Ausgangszellen
zu charakterisieren, werden eingesetzt, um die klonierten Zellen
zu charakterisieren. Der
Karyotyp der Zellen wird an den Metaphasen der Zellen, die gemäß einem
klassischen Protokoll nach einer Behandlung der in Proliferation
befindlichen Zellen mit Colcemid (Puls von einigen Stunden mit 0,05 bis
0,15 μg/ml
Endkonzentration, vorzugsweise 0,08 μg/ml) erhalten worden sind,
analysiert. Die ausgebreiteten Metaphasen werden unter dem Mikroskop
beobachtet und die Anzahl von Makrochromosomen gezählt. Die
Zählung
der Minichromosome ist machbar, aber delikater und eine detailliertere
Auswertung durch in situ-Hybridisierung
kann gleichfalls ins Auge gefasst werden. Außerdem unterstützt die
Verifizierung des Vorhandenseins des Transgens durch FISH (in situ-Hybridisierung
mit einer fluoreszierenden Sonde) die durch Southern-Blot vorgenommenen
Beobachtungen, was die Einmaligkeit des Ereignisses angeht. Der
erste eingesetzte immunzytochemische Test ist der Nachweis der endogenen
alkalische Phosphatase-Aktivität.
Alle in der PCR positiven und durch Southern-Blot Analyse validierten
Klone werden im Vergleich zu den Ausgangszellen untersucht. Wenn
die Ausgangszellen positiv sind, wird das Vorhandensein der für Stammzellen
spezifischen Oberflächenantigene
gleichfalls auf der Ebene der erhaltenen rekombinierten Klone untersucht.
Die Reaktionen mit den Antikörpern
SSEA-1, TEC-01 und EMA-1 werden systematisch ausgeführt. Die
Telomerase-Aktivität
erweist sich als eines der guten Kriterien, um den undifferenzierten
Zustand von Stammzellen zu beurteilen. Auch wird wie bei den Ausgangszellen
die Telomerase-Aktivität
der verschiedenen rekombinierten Klone analysiert. Während der
Passage der Klone zu deren Amplifizierung und Einfrieren wird ein
kleines Aliquot von 5 × 103 bis 5 × 105 Zellen in Form von zellulärem Bodensatz
hergestellt und die Telomerase-Aktivität von diesen
Zellen wird mit Hilfe des Kits „Telo TAGGGTélomerase
PCR Elisa” (Roche) ausgewertet.
Das Niveau der Telomerase-Aktivität scheint eine gute Wiederspiegelung
der Qualität
der Klone insbesondere nach dem durch das Arzneimittel ausgeübten Selektionsdruck
zu sein. Die
nachfolgende Tabelle 9 veranschaulicht einige von diesen Eigenschaften
an rekombinierten Klonen: Die
Zellen werden in Vertiefungen einer Platte mit 12 Vertiefungen zu
im Mittel 104 Zellen pro Vertiefung ausgesät. Die verschiedenen
Charakterisierungen erfolgen nach Fixierung im Falle des AP- und
Antikörper-Nachweises
und an trockenem Bodensatz, der gemäß dem in dem Roche-Kit etablierten
Protokoll lysiert wird, in dem Falle der Telomerase-Aktivität (NT =
nicht getestet). Die
erhaltenen und nach PCR- und Southern-Blot-Analyse selektionierten
Klone sind a priori auf einem einzigen Allel modifiziert (Nachweis
des wilden Allels im Rahmen einer Southern-Blto-Analyse). Die Gewinnung
von zwei modifizierten Allelen kann in vivo und in vitro ausgeführt werden.
In vivo werden die hinsichtlich der Modifizierung homozygoten Tiere
erhalten nach Kreuzung der heterozygoten Tiere, die ihrerseits von chimären Tieren,
die direkt nach Injektion der modifizierten Zellen erhalten werden,
abstammen. In vitro ist die Allelkonversion des zweiten, noch nicht
modifizierten Allels insbesondere möglich durch die Anwendung einer Selektion
mit hohen Dosen des Arzneimittels G418 (0,5 bis 5 mg/ml) an den
Klonen. Dieser Ansatz erlaubt, interessante Mengen von Proteinen
zugleich in vitro als auch in vivo zu erhalten. Das
Ziel der Erfindung besteht darin, die Moleküle in der physiologischen Flüssigkeit
des Tiers, insbesondere im Blut und Eiweiß, zu produzieren. Um diesen
Ansatz zu realisieren, werden die Vektoren, insbesondere die homologen
Rekombinationsvektoren gemäß den zuvor
beschriebenen Schemata kontruiert. Diese Rekombinationsvektoren
steuern zielgerichtet verschiedenen Genorte, darunter hauptsächlich den
Genort des Ovalbumins und jenen des Lysozyms an. Ein wichtiger Punkt
ist die Validierung dieser Vektoren durch einen in vitro-Ansatz.
Insbesondere ist es wichtig, die Faltung des herzustellenden Proteins,
die in vitro in einem Vogel-System erhaltenen posttranslationalen
Modifizierungen und den Einfluss des homozygoten Zustands des modifizierten
Genorts auf die Expression des Moleküls von Interesse zu verfolgen.
Die Sekretionsstrategien werden gleichfalls über ein in vitro-Modell mit
je nach Konstrukt unterschiedlichen Signalpeptiden und Spleißstrategien
untersucht. Es
wurden zwei komplementäre
Systeme entwickelt, um jede der hauptsächlichen Klassen von Vektoren
zu untersuchen. In
einem ersten Ansatz kann die in vitro-Differenzierung der Klone
von Stammzellen mit Erfolg eingesetzt werden, um die Produktion
der Moleküle
im Überstand
der Kulturen zu verfolgen. Die eingesetzten Bedingungen von Medien
sind auf die Bildung von „embryoid
bodies” gerichtet,
die der Anwesenheit von verschiedenen Mitteln, wie Induktoren, wie
beispielsweise Dimethylsulfoxid, Retinsäure, TPA, PMA, LIPS, Faktoren, wie
beispielsweise FGF, TGFβ,
MCSF, cMGF, Hormonen, wie Steroiden, Östradiol, Dexamethason, Progesteron
oder anderen Familien von Hormonen, ausgesetzt werden. Die
LMH-Zellen sind Vogel-Zellen, die von einem hepatischen Tumor stammen.
Nach einigen Beispielen, die der Literatur entnommen werden können, scheint
das Gen des Ovalbumins in der LMH-Linie gemäß unterschiedlichen hormonalen
Stimulationsbedingungen aktivierbar zu sein. Insbesondere erlaubt
eine Kombination von Insulin, Östradiol
und Dexamethason (Steroid, welches die Wirkung der Glucocorticoide
nachahmt) eine mehr oder weniger bedeutende Induktion des Gens des
Ovalbumins. Diese Stimulation kann an den rekombinierten Klonen
der LMH-Zellen eingesetzt werden. Cotransfektionen mit den nukleären Rezeptoren
ER und COUTF werden gleichfalls ins Auge gefasst, um die Konzentration
von diesen Rezeptoren in den Zellen zu erhöhen und so eine bessere Induktion
des Gens des Ovalbumins unter der Wirkung der verschiedenen eingesetzten
Induktoren zu erlauben. Die Vektoren der Familie pOvaRH werden in
diesem Ansatz bevorzugt eingesetzt. Es
erscheint möglich,
die Differenzierung der HD11-Linie, einer Linie von Monozyten vom
Huhn, zu Makrophagenzellen durch verschiedene Behandlungen, darunter
eine Wirkung durch die Lipopolysaccharide, zu induzieren (Goethe
et al., 1994; Goethe und Phi-Van, 1998). In einem Ansatz einer Stimulation
durch diese Substanzen wird der Überstand
der insbesondere mit den Vektoren aus der Familie pLysoRH rekombinierten Klone
untersucht und erlaubt, bedeutende Mengen von Proteinen von Interesse
zu erhalten. Die
nachfolgende Tabelle 10 veranschaulicht diese Produktion von Molekülen von
Interesse ausgehend von mit einem der Vektoren aus der pLysoRH-Familie
rekombinierten HD11-Zellen spezifisch unter der Wirkung von Induktor,
darunter den LPS. Die
HD11-Zellen werden transfiziert und rekombinierte Klone mit dem
Vektor pLysoRH Epo erhalten. An den durch LPS (5 μg/ml) stimulierten
Klonen werden zwei Analysen ausgeführt: Die
Klone von modifizierten Stammzellen werden mit Hilfe der verschiedenen
zuvor beschriebenen Immunzytochemie-Marker wie die Ausgangszellen
charakterisiert. Am Ende von diesen Kontrollen werden bestimmte
Klone eingesetzt, um modifizierte Tiere zu erzeugen, die dazu bestimmt
sind, das Molekül
von Interesse zu produzieren. Unter den verschiedenen Strategien,
die eingesetzt werden, um diese Tiere zu produzieren, kann man unterscheiden: Die
modifizierten Zellen werden in die Keimhöhle eines Empfänger-Embryos
im Blastula-Stadium gemäß einem
zuvor beschriebenen Protokoll (Pain et al., 1996. Die Mit
durch den einfachen Vektor pEGFP-SV40 Néo modifizierten Zellen erhaltene
Chimäre. Die
auf biastodermalem Wege injizierten Eier werden in verschiedenen
Stadien geöffnet
und die Embryos entnommen, um analysiert zu werden. Die extrahierte
DNA wird einer genomischen PCR-Reaktion mit verschiedenen Oligonukleotiden
unterzogen, um das Vorhandensein des Transgens (hier E-GFP) nachzuweisen.
Der obere Abschnitt des Gels zeigt so 21 analysierte Proben, darunter
unter den 15 analysierten injizierten Embryos 11 positiv
detektierte. Der untere Abschnitt zeigt die Analyse, die mit einem
anderen Paar von Oligonukleotiden, welches erlaubt, die Qualität der DNA
und der Extraktion zu validieren, ausgeführt worden ist. Die
nachfolgende Tabelle 11 veranschaulicht die Gewinnung von Chimären mit
einem rekombinierten Klon. Die
DNA der lebenden chimären
Tiere wurde aus einer Gefiederbiopsie extrahiert und ein PCR-Nachweis
bestätigt
das Vorhandensein des Transgens in den Proben (Ergebnis nicht gezeigt). Die
modifizierten Zellen werden in den Blutkreislauf eines Embryos mit
48 bis 72 h Bebrütung über den intrakardialen
Weg injiziert. In diesem Entwicklungsstadium erlaubt die Morphogenese
des Embryos eine bedeutende Zugänglichkeit
der in Bildung befindlichen Herzhöhle. Mit Hilfe von feinen Kapillaren
werden die Zellen direkt injiziert. Die Entwicklung geht normalerweise
ihren Gang bis zum Schlüpfen. Die
nachfolgende Tabelle 12 veranschaulicht den phänotypischen Beitrag von modifizierten
Klonen in den Geweben von Embryos durch intrakardiale Injektion. Die
Zellen eines mit einem einfachen Vektor modifizierten Klons (in
dem Beispiel wird der Klon 14A mit dem Vektor pWS65, welcher pCMV
E-GFP SV40 Néo
enthält,
erhalten) oder eines mit einem Rekombinationsvekfor modifizierten
Klons werden intrakardial direkt in den Blutkreislauf eines einige
Stunden (36 bis 72 h) bebrüteten
Embryos injiziert. In dem Beispiel werden die Gewebe je nach Fall
Embryos von 5 bis 21 Tage entnommen. Eine genomische PCR erlaubt,
das Vorhandensein des Transgens nachzuweisen, wohingegen eine andere
PCR an einem endogenen Gen die Qualität der analysierten DNA validiert. Die
modifizierten Zellen sind Quellen für modifizierte Kerne, die im
Rahmen von nukleären
Transfertechniken in eine Empfänger-Oocyte
eingesetzt werden können,
um einen Embryo zu rekonstituieren. In diesem Falle trägt das Tier
in seinem genetischen Erbgut direkt die durch den Kern beigetragene
Modifizierung und diese wird folglich an alle seine Nachkommen weitergegeben.
Die aktuellen Fusionstechniken der Zelle und/oder allein des Kerns
mit der vorbereiteten Oocyte erlauben, einen solchen Ansatz ins
Auge zu fassen. Die Aktivierungsphasen erlauben dann eine wirksame
Reprogrammierung des Donor-Kerns anstelle des haploiden Genmaterials
der Empfänger-Oocyte. Verfahren zur Gewinnung einer embryonalen Stammzelle
(ES) von Vögeln,
welche durch homologe Rekombination modifiziert ist, wobei das Verfahren
die folgenden Schritte umfasst: Verfahren nach Anspruch 1, dadurch gekennzeichnet,
dass der Vektor d) die das exogene Protein kodierende Sequenz fusioniert
an ihrem 5'-Ende
mit c) einer kurzen Intron-Sequenz,
umfassend insbesondere die Sequenz SEQ ID No. 1, welche ihrerseits
fusioniert ist mit b) einer Peptidsekretionssignalsequenz, insbesondere
der das Signalpeptid von Lysozym kodierenden Sequenz, welche die
Sequenz SEQ ID No. 2 umfasst, umfasst. Verfahren nach einem der Ansprüche 1 und 2, dadurch gekennzeichnet,
dass der Vektor d) die das exogene Protein kodierende Sequenz fusioniert
an ihrem 3'-Ende
mit einer poly A-Sequenz
umfasst. Verfahren nach einem der Ansprüche 1 bis 3, dadurch gekennzeichnet,
dass der Vektor mindestens eine interne Ribosomeneintrittssequenz
IRES fusioniert mit mindestens zwei Sequenzen, welche das exogene
Protein von Interesse kodieren, umfasst. Verfahren nach einem der Ansprüche 1 bis 4, dadurch gekennzeichnet,
dass der Vektor mindestens eine IRES-Sequenz fusioniert mit mindestens
zwei Sequenzen, welche unterschiedliche Ketten, welche ein Protein von
Interesse bilden, insbesondere die schwere und die leichte Kette
eines Antikörpers
unabhängig
von dessen Natur, insbesondere einen monoklonalen Antikörper, ein
fab-Fragment, kodieren, umfasst. Verfahren nach einem der Ansprüche 4 und 5, dadurch gekennzeichnet,
dass die IRES-Sequenz
aus der Gruppe der IRES-Sequenzen der Gruppe I oder der Gruppe II,
insbesondere den Sequenzen V130, Idemfix, Zam ausgewählt wird. Verfahren nach einem der Ansprüche 1 bis 6, dadurch gekennzeichnet,
dass in Schritt A) das Transfektionsverfahren unter den Transfektionsverfahren
mit Hilfe eines Liposoms, eines Polykations oder durch Elektroporation
ausgewählt
wird. Verfahren nach Anspruch 7, dadurch gekennzeichnet,
dass in Schritt A) das Transfektionsverfahren unter den Verfahren
durch Elektroporation ausgewählt
wird. Verfahren nach einem der Ansprüche 1 bis 8, dadurch gekennzeichnet,
dass die Zellen ES-Zellen von Vögeln
sind, welche aus dem Inkulturnehmen von Blastodermen stammen. Verfahren nach einem der Ansprüche 1 bis 9, dadurch gekennzeichnet,
dass die Zellen ES-Zellen von Vögeln
sind, die spezifisch mit mindestens einem unter ECMA-7, SSEA-1,
SSEA-3, TEC-01, EMA-1 und EMA-6 ausgewählten Antikörper reagieren. Verfahren nach einem der Ansprüche 1 bis 10, dadurch gekennzeichnet,
dass der Kulturüberstand
der rekombinierten Klone das exogene Protein von Interesse enthält, insbesondere
nach Induktion des Klons mit Hilfe von verschiedenen Induktoren,
insbesondere Retinsäure,
Dimethylsulfoxid, TPA oder speziellen Kulturbedingungen, insbesondere
durch die Bildung von „embryoid
bodies”. Verfahren nach einem der Ansprüche 1 bis 11, dadurch gekennzeichnet,
dass die beiden Allele des Ziel-Genorts modifiziert sind. Verfahren nach einem der Ansprüche 1 bis 12, dadurch gekennzeichnet,
dass die ES-Zellen
Zellen sind, die einen alkalische Phosphatase-positiven Phänotyp aufweisen. Verfahren nach einem der Ansprüche 1 bis 13, dadurch gekennzeichnet,
dass das eingesetzte Medium ein Zytokin, welches in der aus LIF,
IL-11, IL-6 und deren verschiedenen Mischungen gebildeten Gruppe
ausgewählt
wird, umfasst. Verfahren nach einem der Ansprüche 1 bis 14, dadurch gekennzeichnet,
dass das eingesetzte Medium verschiedene Faktoren, insbesondere
SCF, IGF-1, bFGF, CNTF und Oncostatin, umfasst. Verfahren zur Herstellung eines Proteins von Interesse,
umfassend die Extraktion des in den Überstand der Zellen, die aus
dem Verfahren nach einem der Ansprüche 1 bis 15 stammen, exprimierten
exogenen Proteins. Verwendung einer Vogel-Zelle, die durch ein Verfahren
nach einem der Ansprüche
1 bis 15 erhalten worden ist, für
die Herstellung des exogenen Proteins von Interesse. Verfahren zur Gewinnung eines Tiers, das zu der
Spezies der Vögel
gehört,
welches in der Lage ist, ein exogenes Protein von Interesse zu exprimieren,
dadurch gekennzeichnet, dass es die folgenden Schritte umfasst: Verfahren zur Gewinnung eines Tiers, das zu der
Spezies gehört,
nach Anspruch 18, dadurch gekennzeichnet, dass der in Schritt a)
zur Gewinnung von modifizierten Vogel-Zellen eingesetzte Vektor
eine gewebespezifische Expression erlaubt. Verfahren zur Gewinnung eines Tiers nach Anspruch
19, dadurch gekennzeichnet, dass der in Schritt a) eingesetzte Vektor
eine gewebespezifische Expression des exogenen Proteins in der Leber,
im Blut, im Knochenmark, in den lymphatischen Organen oder dem Eileiter
erlaubt. Verfahren zur Herstellung eines Proteins von Interesse,
umfassend die Extraktion des in den Geweben eines Tiers, das ausgehend
von dem Verfahren nach einem der Ansprüche 18 bis 20 erhalten worden
ist, exprimierten exogenen Proteins. Verfahren nach Anspruch 21, dadurch gekennzeichnet,
dass das Protein aus dem Blut, dem Eigelb oder dem Eiweiß extrahiert
wird. Ei, welches ausgehend von einem durch ein Verfahren
nach einem der Ansprüche
18 bis 20 erhaltenen Tier erhalten werden kann, dadurch gekennzeichnet,
dass ein Teil des Ovalbumins oder des Lysozyms teilweise oder vollständig durch
das exogene Protein von Interesse ersetzt ist. Ei nach Anspruch 23, dadurch gekennzeichnet, dass
das exogene Protein von Interesse unter den Peptiden von therapeutischem
Interesse, den Interleukinen, den Zytokinen, den Hormonen und den
Antikörpern
ausgewählt
wird.TECHNISCHES GEBIET:
STAND DER TECHNIK:
ZUSAMMENFASSUNG DER ERFINDUNG:
ein Verfahren zur Gewinnung
einer embryonalen Stammzelle (ES) von Vögeln, welche durch homologe
Rekombination modifiziert ist, wobei das Verfahren die folgenden
Schritte umfasst:
ein
Verfahren zur Herstellung eines Proteins von Interesse, umfassend
die Extraktion des in den Überstand der
Zellen, die aus dem Verfahren nach einem der Ansprüche 1 bis
15 stammen, exprimierten exogenen Proteins.
dadurch
gekennzeichnet, dass:
der eine homologe Rekombination ermöglichende
Vektor, der in Schritt A) eingeführt
wird, in einer Plasmid-Grundstruktur eine Aneinanderreihung von
mindestens einem Element umfasst, welches nacheinander ausgewählt wird
unter:
das dadurch gekennzeichnet ist, dass:
der Genort,
der zielgerichtet durch den Rekombinationsvektor angesteuert wird,
der Genort von Ovalbumin oder der Genort von Lysozym ist.
eine Verwendung
einer Vogel-Zelle, die durch ein Verfahren nach einem der Ansprüche 1 bis
15 erhalten worden ist, für
die Herstellung des exogenen Proteins von Interesse.
ein Verfahren
zur Gewinnung eines Tiers, das zu der Spezies der Vögel gehört, welches
in der Lage ist, ein exogenes Protein von Interesse zu exprimieren,
dadurch gekennzeichnet, dass es die folgenden Schritte umfasst:
ein Verfahren zur
Herstellung eines Proteins von Interesse, umfassend die Extraktion
des in den Geweben eines Tiers, das ausgehend von dem Verfahren
nach einem der Ansprüche
18 bis 20 erhalten worden ist, exprimierten exogenen Proteins.
ein
Verfahren nach Anspruch 21, das dadurch gekennzeichnet ist, dass
das Protein aus dem Blut, dem Eigelb oder dem Eiweiß extrahiert
wird.
ein Ei, welches ausgehend von einem durch ein Verfahren
nach einem der Ansprüche
18 bis 20 erhaltenen Tier erhalten werden kann, welches dadurch
gekennzeichnet ist, dass ein Teil des Ovalbumins oder des Lysozyms
teilweise oder vollständig
durch das exogene Protein von Interesse ersetzt ist.BESCHREIBUNG
I. Verschiedene Vektoren können für die Transformation
der Zellen eingesetzt werden:
1.1 – Rekombinationsvektoren.
Unter endogenem Protein von Interesse
versteht man ein jegliches in dem System, insbesondere dem Ei, vorhandenes
Protein, das in variablen, aber in einer normalen physiologischen
Situation nachweisbaren Konzentrationen vorhanden ist.Länge der homologen Fragmente
Zu diesem Zweck umfasst der Vektor heterologe
Segmente:
Induktion eines zielgerichteten Schnitts
Linearisierung eines Vektors
Konditionelle Struktur der Expressionsvektoren
Grundelemente der Konstrukte
1.2 Einfacher Expressionsvektor (Vergleichsbeispiel)
Abkürzungen
Beispiel 1: Das Ei als Produktionssystem
1.1 Die Proteine des Eiweiß
Ganzes
Ei Eiweiß Eigelb Wasser 40 31 9 Trockensubstanz 20 4 10 Proteine 6,9 3,6 3,2 Lipide 6,4 < 0,2 6,4 Glucide 0,2 0,2 < 0,1 Protein %
des Proteins MG pl %
Kohlen hydr. (carb.) Funktion AS S-S Ovalbumin 54 45,0 4,5 3,5 Strukturell 385 1 Conalbumin 12–13 77,7 6,0 2,6 Eisentransport 686 15 Ovomucoid 11 28,0 4,1 16 Proteinaseinhibitor 185 9 Ovomucin a
Ovomucin b 1,5–3,5 210
7204,5 13
58 Strukturell 1276
246Lysozym 3,5 14,3 10,7 0 Proteolytisches
Enzym 129 4 Ovoinhibitor 0,5–1,5 49,0 5,1 5–9,6 Proteinaseinhibitor 21 Ovoglycoprotein 0,5–1,0 24,4 3,9 ? Ovoflavoprotein 0,8 32,0 4,0 11 Riboflavintransport 234 9 Ovostatin 0,5 780 4,9 5,8 Proteinaseinhibitor 2762 Ovoglobulin
G2 > 1 47 4,9 ? ? 102 Ovoglobulin
G3 > 1 50 4,8 ? ? 103 Cystatin 0,05 12,7 5,1 0 Proteinaseinhibitor 124 2 Avidin 0,05 68,3 10 7 Biotintransport 4 × 129 4 × 1 Thiamin bindendes Protein < 0,05 38 0 Thiamintransport Andere Proteine 9 1.2 Die Komponenten des Eigelbs
Beispiel 2: Beschreibung der molekularen
Genorte und deren transkriptionelle Regulation
cDNA Gen Gen
(partiell) Ovalbumin V00383 J00895 Ovalbumin
X J00917
5' ex 1 (Exon 1), J00918
ex5, J00919 ex6, J00920 ex7 Ovalbumin
Y J00922 Conalbumin X02009 Y00407 Ovomucoid J00902 J00897
(5'), J00898 (3', kodierend), J00899
(3', nicht-kodierend) Ovomucin ☐ AB046524 Lysozym X61198 X98408
X00589
X00591
X05461
X05463
X05462J00882
5' ex1
J00883
ex2
J00884 ex3
J00885 ex4 3'Ovoinhibitor
(16 Exons) M15962
ex1, M16127 ex2,
M16128 ex3
M16129 ex4, M 16130 ex5,
M16131
ex6
M16132 ex7, M16133 ex8,
M16134 ex9
M16135 ex10,
M16136 ex11,
M16137 ex12
M16138 ex13, M16139 ex14,
M16140
ex15
M16141 ex16Ovostatin X78801 Cystatin J05077 M95725 X62413
(synthetische Mut. V55D)
X62411 (synthetische Mut. Q53N S56A)
X62412
(synthetische Mut. R52L Q53E)
X62409 (synthetische Mut. Q53N)
X62408
(synthetische Mut. Q53E)
X62407 (synthetische Mut. L54M G57A)
X62410
(synthetische Mut. Q53N G57C)
X62406 (synthetische Mut. G57A)
X14685
(1-23 SIM 129 189)Avidin
(Avd) X05343 AJ311647 Avr1 AJ311647 221611 Avr2 AJ311648 221554 Avr3 221612 Avr4 222883 Avr5 222882 Avro AJ237658 Avr7 AJ237659 2.1 Genort des Ovalbumins
2.2 Der Genort des Lysozyms
2.3 Der Genort des Conalbumins
Beispiel 3: Vereinfachende äußere Elemente
Bedingungen 1 2 3 4 5 Vergleichsprobe ppréOva RH (μg) 5 5 5 5 5 0 pMCS5
(μg) 5 4 3 2 0 10 pRad54
(μg) 0 1 2 3 5 0 Fugène 30 30 30 30 30 30 DMEM 970 970 970 970 970 970 Anzahl von
Klonen Schale
1 344 304 172 248 292 0 Schale
2 128 324 212 264 300 0 Mittelwert 236 314 193 256 296 0 Beispiel 4: Wahl des Signalpeptids
Beispiel 5: Konstruktion von Rekombinationsvektoren
5.1 Gewinnung der intermediären Elemente
– Kassette
pCMV Néo
polyA
– Herstellung
der cDNAs von Interesse
5.2 Gewinnung der genomischen Fragmente
5.3. Konstruktion des pOvaRH-Gerüsts
5.4 Konstruktion des pLysoRH-Gerüsts
5.5 Anwendung für die Expression von verschiedenen
Proteinen
Beispiel 6: Transfektion von verschiedenen
Vogel-Zellen mit den Vektoren
6.1 Transfektionsmethoden
– die Elektroporation
Kapazitanz (μF) 25 150 300 500 700 1000 Klone
A 5 122 226 335 408 286 Klone
B 9 149 297 454 443 361 Klone
C 10 180 218 361 402 365 Mittelwert 8 150 247 383 418 337 Standardabweichung 3 29 43 63 22 45 – die Liposome
Tabelle 6: Transfektion von verschiedenen
Zellarten mit Rekombinationsvektoren
Zellen
(Nr. der Passage) Vektoren Analysierte
Klone Anhand
positiv von
PCR e Klone nb nb % LMH
(p25) pOvaRH
Hélio 35 1 3,8 HDl
1 pLysoRH
GFP 40 1 2,5 HD11 pLysoRH
Epo 36 3 8,3 Valo4
(p39) pOvaRH
Hélio 64 1 1,6 Valo4
(p53) pOvaRH
LacZ 28 1 3,6 S86N
48 (p13) pOvaRH
Hélio 48 1 2,1 S86N
16 (p123) pOvaRH
Hélio 264 23 8,7 S86N
16 (p129) pOvaRH
LacZ 168 1 0,6 S86N
16 (p129) pOvaRH
hélio 157 14 8,9 S86N
16 (p137) pOvaRH
Hélio 96 3 3,1 S86N
16 (p140) pOvaRH
LacZ 143 7 4,9 S86N
16 (p203) pLysoRHx 94 25 26 S86N
45 (p71) pLysoRHx 94 10 10,4 S86N
45 (p79) pLysoRH
Hélio 96 15 15,6 S86N
88 (p14° pOvaRH
LacZ 79' 1 1.2 Valo8
(p10) pOvaRH
Hélio 35 1 2.8 Valo8
(p 13) pOvaRH
Hélio 120 3 2.5 6.2 Effizienz hinsichtlich stabilen Transfektionen
mit den Rekombinationsvektoren
6.3 Eingesetzte Zellen
6.3.1 Embryonale Fibroblasten vom Huhn
(CEFs) (Vergleichsbeispiel)
6.3.2 Linie LMH (Vergleichsbeispiel)
6.3.4 Embryonale Stammzellen
Tabelle 7: Transfektion von CECs-Zellen
mit verschiedenen einfachen Expressionvektoren
Versuche 1 2 3 4 5 6 Mittelwert Standard abweichung Anzahl von 75 90 135 124 146 106 113 27 Klonen 6.4 Physiologischer Zustand der Zellen
6.5 Klonierung und Nachweis der Rekombinationsereignisse
6.5.1 Klonierung
6.5.2 Screening der Klone durch PCR
Legenden Fig. 6: Nachweis
der positiven rekombinanten Klone durch PCR
Legende der Fig. 7. Nachweis
des Geschlechts von verschiedenen identifizierten, anhand von PCR
positiven Klonen
6.5.3 Southern Blot
die Größen des
zielgerichtet angesteuerten Genorts erhalten geblieben sind und
dass es keine bedeutendere Deletion gibt,
ein einziges Rekombinationsereignis
stattgefunden hat und dass das Genom keine anderen zufälligen Integrationen
enthält.Tabelle 8: Nachweis von Rekombinationsereignissen
durch Southern-Blot
Verdau BamHI Scal Sonde -RH RH
Hélio –RH RH
Hélio Ovalbumin
3' 18,5
kb 7
kb 12
kb 12,7
kb Ovalbumin
5' 18,5 9,5 12 12,7 Neomycin / 2 / 12 6.6 Charakterisierung der Klone
6.6.1 Verifizierung des Karyotyps
6.6.2 Endogene alkalische Phosphatase-Aktivität
6.6.3 Oberflächenantikörper
6.6.4 Telomerase-Aktivität
Zellen AP-Nachweis* Antikörper TEC01 (%) Antikörper EMA-1 (%) Telomerase
(Leerwert = 0,07) –RA +RA –RA +RA -RA +RA –RA +RA S86N16* ++++ – 90 < 10 90 10 > 1,5 < 0,2 C10 +++ – 70 < 10 90 < 10 1,23 0,282 C44 +++ – 70 < 10 90 < 10 1,13 0,20 D21 +++ – 90 < 10 90 < 10 1,04 0,15 A5-1 NT NT NT NT NT NT 1,08 0,257 A21 +++ + NT NT NT NT 1,13 0,62 A
37 +++ +/– NT NT NT NT 1,09 0,566 6.7 Allelkonserversion
Beispiel 7: In vitro-Test der Rekombinationsvektoren
hinsichtlich der Produktion der Proteine von Interesse
7.1 In vitro-Differenzierung der rekombinierten
Klone
7.2 Stimulation der Zellen in rekombinierten
Linien
7.2.1 Validierung der pOvaRH-Vektoren
7.2.2 Validierung der pLysoRH-Vektoren
Tabelle 10: Stimulation von mit dem Vektor
pLysoRH Epo tranfizierten HD11-Zellen
O.
D. bei 450 Konzentration Standard S1 0 0 S2 0,12 11 S3 0,26 20 S4 0,46 41 S5 0,86 86 S6 2,00 179 Positive
Kontrolle 0,79 71,4 Negative
Kontrolle 0,09 1,2 HD11-Ausgangszellen –LPS 0 <0,5 Nicht-rekombinierte
Klone +LPS 0 <0,5 Klon 1 pLysoRH Epo –LPS 0,67 61,3 +LPS 2,22 199 Beispiel 8: Injektion von modifizierten
Zellen in Empfänger-Embryos
und Gewinnung von Tieren
8.1 Intrablastodermale Injektion
Legende der Fig. 9: Gewinnunq
von Chimären
mit den modifizierten Klonen
Injizierte
Zellen Vektor Lebende
Tiere Chimäre (%) S86N
66(p5)* x 13 1
(8%) S86N
48 Klon A5 pOvaRH
Hélio 37 1
(3%) S86N
48 Klon A5 pOvaRH
hélio 43 3
(7%)** 8.2. Intrakardiale Injektion
Tabelle 12: Gewinnung von phänotypischen
Chimären
durch intrakardiale Infektion
Zellen Embryos
mit Injektion Analysierte
Embryos Positive
Embryos % Bsp.
1 40 16 2 12,5 Bsp.
2 40 18 3 16,7 Bsp.
3* 50 14 2 14,3 Bsp.
4* 50 18 2 11,1 Bsp.
5* 50 22 1 4,5 8.3 Verwendung der modifizierten Kerne
als Quelle für
Kerne in einem nukleären
Transferverfahren (Vergleichsbeispiel)
REFERENZEN
A) die Einführung eines eine homologe Rekombination
ermöglichenden
Vektors in die ES-Zelle
von Vögeln durch
ein Transfektionsverfahren;
B) die Selektion der Zellen durch
die Zugabe eines Selektionsmittels in das Kulturmedium; und
C)
das Screenen der resistenten Klone und Amplifizierung,
dadurch
gekennzeichnet, dass:
der eine homologe Rekombination ermöglichende
Vektor, der in Schritt A) eingeführt
wird, in einer Plasmid-Grundstruktur eine Aneinanderreihung von
mindestens einem Element umfasst, welches nacheinander ausgewählt wird
unter:
a) einem genomischen DNA-Fragment, enthaltend den 5' gelegenen Homologie-Sequenzabschnitt
des Zielgens, fusioniert mit
b) einer Sekretionssignal-Nukleotidsequenz,
fusioniert mit
c) einer kurzen Intron-Nukleotidsequenz, fusioniert
mit
d) einer Nukleotidsequenz, welche ein exogenes Protein
von Interesse kodiert, fusioniert mit
e) einer poly A-Transkriptionsterminationssequenz,
fusioniert mit
f) einer eine positive Selektion ermöglichenden
Kassette, umfassend einen Promotor, ein Resistenz gegen ein Selektionsmittel
verleihendes Gen und eine poly A-Transkriptionsterminationssequenz,
wobei die Kassette fusioniert sein kann mit
g) einem genomischen
DNA-Fragment, welches den 3' gelegenen
Homologie-Sequenzabschnitt
des Zielgens enthält,
h)
einer eine negative Selektion ermöglichenden Kassette, umfassend
einen Promotor, ein Gen, welches die Umwandlung eines in dem Kulturmedium
vorhandenen Substrats zu einer für
die Zelle, die das Gen exprimiert, toxischen Substanz sicherstellt,
und eine poly A-Transkriptionsterminationssequenz
und
dadurch gekennzeichnet, dass:
der Genort, der zielgerichtet
durch den Rekombinationsvektor angesteuert wird, der Genort von
Ovalbumin oder der Genort von Lysozym ist.
a)
Gewinnung von Vogel-Zellen, die durch das gemäß einem der Ansprüche 1 bis
15 definierte Verfahren modifiziert worden sind,
b) Einführung der
in Schritt a) erhaltenen Zelle in die Keimhöhle eines Embryos oder in den
Blutkreislauf des Embryos und
c) Inkubation des in Schritt
b) erhaltenen Embryos.